Resumen de la tecnología
Consideraciones sobre el ensayo
Métodos de cuantificación
Equipos y suministros
Equipos & Suministros
Guía de selección de mezclas de PCR Protocolo
Solución_de_problemas
Materiales
Referencias
Descripción general de la tecnología: SYBR Green qPCR
Con el desarrollo de termocicladores que incorporan detección fluorescente, la PCR tiene una aplicación nueva e innovadora. En la PCR rutinaria, el resultado crítico es la cantidad final de amplicón generado tras el proceso. La PCR en tiempo real o cuantitativa y la RT-PCR utilizan la linealidad de la amplificación del ADN para determinar las cantidades absolutas o relativas de una secuencia conocida en una muestra. Mediante el uso de un reportero fluorescente en la reacción, es posible medir la generación de ADN.
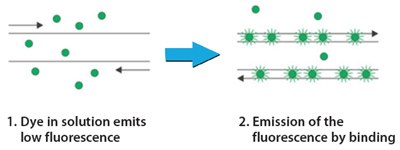
Figura 1.Visión general de la tecnología: SYBR Green qPCR
En la PCR cuantitativa, la amplificación del ADN se monitoriza en cada ciclo de la PCR. Cuando el ADN se encuentra en la fase logarítmica lineal de amplificación, la cantidad de fluorescencia aumenta por encima del fondo. El punto en el que la fluorescencia se vuelve medible se denomina ciclo de cuantificación (Cq) o punto de cruce. Utilizando diluciones múltiples de una cantidad conocida de ADN estándar, se puede generar una curva estándar de concentración logarítmica frente a Cq. La cantidad de ADN o ADNc en una muestra desconocida puede calcularse a partir de su valor de Cq.
A) Las diferentes fases de la reacción:
Baseline: La concentración inicial de molde es baja; por lo tanto, la intensidad de fluorescencia es demasiado baja para ser detectada y sólo es evidente la señal de fondo.
Exponencial: Una vez que el rendimiento del blanco ha alcanzado el umbral de detección, que se muestra como la línea roja de umbral, puede seguirse el curso de la reacción a través de la fase exponencial.
Lineal: A medida que aumenta la concentración de molde, la concentración de ADN polimerasa disponible se reduce y la velocidad de reacción disminuye.
Plateau: No hay suficiente enzima libre para continuar la amplificación, por lo que después de este punto, la reacción se encuentra en el rendimiento máximo, o en la fase plateau.
B)
Las reacciones individuales se caracterizan por el ciclo en el que la fluorescencia se eleva por primera vez por encima del umbral, que se denomina ciclo de cuantificación (Cq). Si el material de partida es abundante, la amplificación se observa en ciclos más tempranos, y el Cq es más bajo. Si el material de partida es escaso, la amplificación se observa en ciclos más tardíos y el Cq es mayor. Esta correlación entre fluorescencia, Cq y cantidad de producto amplificado permite la cuantificación del molde en un amplio rango dinámico.
La PCR en tiempo real también se presta a estudios relativos. Puede realizarse una reacción utilizando cebadores exclusivos para cada región a amplificar y marcados con diferentes colorantes fluorescentes. Varios termocicladores cuantitativos disponibles en el mercado incluyen múltiples canales de detección. En este sistema multiplex, la cantidad de ADN/ADN diana puede compararse con la cantidad de una secuencia de mantenimiento, por ejemplo GAPDH o β-actina.
NR= no recomendado
X= recomendado
XX= método preferido
Consideraciones sobre el ensayo
Preparación del ADN
El paso más importante para garantizar el éxito de la PCR es la preparación de ADN de alta calidad. La integridad y pureza del ADN molde son esenciales. La PCR cuantitativa implica múltiples rondas de reacciones enzimáticas y, por lo tanto, es más sensible a impurezas como proteínas, fenol/cloroformo, sales, EDTA y otros disolventes químicos. Los contaminantes también pueden interferir en la detección por fluorescencia. La relación de los valores de absorbancia a 260 nM y 280 nM da una estimación de la pureza del ADN. El ADN puro tiene una relación A260/A280 de 1,8-2,0. Una relación inferior indica la presencia de contaminantes. Ratios inferiores indican la presencia de contaminantes como proteínas.
Template
Se necesitan muy pocas copias de ácido nucleico diana (equivalente a unos 100 pg de gDNA o cDNA) para iniciar la qPCR. Para minimizar la contaminación con inhibidores de la reacción, la cantidad de molde inicial debe mantenerse en el mínimo necesario para lograr una cuantificación precisa. Cuando el material de partida es ARN, el diseño del cebador y el tratamiento con DNasa I reducirán las señales que puedan generarse a partir de la contaminación por ADNg.
Diseño del cebador
Ya sea que se utilice un colorante de unión a ADNds o una química de detección basada en sondas, el diseño de cebadores de alta calidad es uno de los pasos preexperimentales más cruciales en la qPCR. Los cebadores específicos para la PCR deben diseñarse con la ayuda de un software de diseño de cebadores para eliminar las complicaciones introducidas por los cebadores-dímeros y las estructuras secundarias. Las concentraciones más bajas de cebadores disminuyen la acumulación de la formación de dímeros de cebadores y la formación de productos inespecíficos, lo cual es fundamental al utilizar el colorante SYBR Green I en la PCR cuantitativa.
dNTPs
Las mezclas maestras estándar de PCR/qPCR contienen dATP, dCTP, dGTP y dTTP. Sin embargo, existen algunas mezclas que sustituyen el dTTP por dUTP. Los productos de reacciones anteriores realizadas con dUTP contendrán uracilo en lugar de timina. Estos son susceptibles de ser cortados por la Uracil-DNA-Glicosilasa (UNG). Por lo tanto, la incubación previa de las reacciones posteriores con UNG evita la contaminación por arrastre entre reacciones. Para ser eficaces, todas las reacciones del laboratorio deben utilizar dUTP.
Concentración de magnesio
El cloruro de magnesio (MgCl2) es necesario para la actividad de la transcriptasa inversa, la Taq ADN polimerasa y la Taq ADN 5' a 3' exonucleasa. Las concentraciones óptimas de Mg2+ para reacciones que contienen DLP suelen estar entre 3 - 6 mM. Las concentraciones más bajas de cloruro de magnesio suelen dar lugar a la formación de menos productos inespecíficos. Algunas soluciones ReadyMix se suministran con una concentración 2X de cloruro de magnesio 7 mM (concentración final 3,5 mM). En algunos casos, se suministra un vial de una solución de cloruro de magnesio 25 mM para una mayor optimización de la concentración final de cloruro de magnesio si es necesario. En ocasiones puede ser necesaria una mezcla de reacción que no contenga MgCl2 para poder utilizar una concentración baja, por ejemplo, cuando se utiliza la detección Scorpion Probe.
Transcriptasa inversa
Una enzima transcriptasa inversa que proporcione altos rendimientos de ADNc, al tiempo que retiene la actividad a alta temperatura, es fundamental para el éxito de la RT-qPCR. El rendimiento a altas temperaturas ayuda a garantizar que las regiones de ARN con una estructura secundaria significativa se desestabilicen y sean accesibles para la hibridación y la posterior amplificación. Cuando se realiza la RT-qPCR en un paso, el rendimiento a altas temperaturas permite el uso de cebadores específicos de genes con altas temperaturas de fusión (Tm), lo que aumenta la especificidad de la reacción. Cuando se realizan protocolos de dos pasos, es importante asegurarse de que la enzima produce un rendimiento lineal y proporcional de ADNc a partir de ARN. Minimizar el pipeteo puede disminuir la variabilidad. Algunas ReadyMixes contienen cebadores y otros reactivos necesarios para realizar la RT, por ejemplo, ReadyScript® cDNA Synthesis Mix (RDRT).
Taq ADN Polimerasa
Al igual que ocurre con la selección de la transcriptasa inversa más adecuada para la RT, la selección de la enzima adecuada es vital. Un problema fundamental con la Taq ADN polimerasa natural es que la enzima tiene actividad residual a baja temperatura. La unión inespecífica del cebador conduce a la formación de productos inespecíficos como resultado de esta actividad residual de la polimerasa. Las ADN polimerasas Taq bloqueadas por anticuerpos o químicamente ("arranque en caliente") ayudan a rectificar esta situación impidiendo la actividad de la enzima hasta que comienza el paso de desnaturalización a alta temperatura. Consulte la Guía de selección de mezclas de PCR para definir la mejor polimerasa de arranque en caliente para su aplicación.
Tinte de referencia interno por tipo de instrumento
Algunos termocicladores de PCR en tiempo real requieren un tinte de carga como ROX para controlar la variabilidad en el sistema óptico y normalizar las diferencias en la intensidad de la señal. Asimismo, algunos termocicladores requieren fluoresceína para crear un fondo virtual cuando se trabaja con ensayos de tinte SYBR Green I (que tienen un fondo muy bajo). Estos pueden suministrarse en el ReadyMix o como componentes separados para que pueda utilizarse la concentración adecuada. En algunos casos, se incluye un vial de colorante de referencia interna para la normalización de la reacción. La excitación máxima de este colorante es de 586 nM y la emisión máxima es de 605 nM. Los ajustes estándar del instrumento para el colorante de referencia ROX son satisfactorios para la medición del colorante de referencia interno. Este colorante de referencia interna es necesario para los sistemas de detección de secuencias ABI.
Instrumentos
Será necesario seleccionar los reactivos compatibles con los instrumentos. Las plataformas utilizan diferentes colorantes de normalización, por lo que será necesario seleccionar reactivos con colorantes de normalización compatibles (consulte Apéndice 1).
Muchos instrumentos de qPCR se han diseñado para admitir una gama específica de aplicaciones, por ejemplo, contraste la capacidad de alto rendimiento del ABI 7900 que utiliza la carga automática de placas de 384 pocillos con el instrumento Illumina Eco que admite una sola placa de 48 pocillos. El instrumento más adecuado responde a las necesidades de la investigación. Es deseable seleccionar un instrumento con un software fácil de usar que realice las funciones más deseables y tenga flexibilidad en cuanto a la salida de datos para que puedan manipularse fácilmente en paquetes de software de análisis estadístico posteriores. Esto reduce el tiempo necesario para formar al personal y, por tanto, para empezar a generar resultados. Las características adicionales que se requieren incluyen un bloque de PCR que sea absolutamente uniforme (una desviación máxima absoluta de 1Cq = 2 veces a través de 96 pocillos de replicación) y un sistema óptico que excite y detecte la emisión de la forma más sensible y uniforme posible a través de una amplia gama de longitudes de onda. Esto permite una amplia selección de fluoróforos y posibilita la multiplexación. Otras características a tener en cuenta son los costes operativos asociados a consumibles específicos, por ejemplo, si no se utiliza una placa de microtitulación estándar para las reacciones y también la conveniencia de cargar placas/tubos que tengan un formato no estándar.
Controles
Un control positivo siempre es útil para asegurarse de que todos los componentes del kit funcionan correctamente. Un control sin plantilla/negativo es necesario para determinar si hay contaminación. Una señal en el control sin molde demuestra la presencia de contaminación de ADN o la formación de dímeros de cebadores.
Tampón
Los tampones o mezclas maestras de reacción suelen contener dNTPs, una Taq ADN polimerasa, MgCl2 y estabilizadores. También pueden incluirse SYBR Green I, ROX™, fluoresceína y colorantes de carga inertes, dependiendo de la química de detección, el instrumento y los requisitos de la reacción. Los componentes del tampón de PCR y los estabilizadores suelen ser propiedad del fabricante. Si se compran por separado, la flexibilidad es máxima, ya que cada ingrediente puede optimizarse individualmente en la reacción. Sin embargo, por el contrario, si bien la compra de los ingredientes juntos como mastermix reduce la flexibilidad, aumenta la consistencia del lote y la comodidad al tiempo que reduce el número de pasos de pipeteo y, por lo tanto, las posibilidades de error y contaminación.
Análisis de datos
Siga las recomendaciones del instrumento de tiempo real utilizado para realizar la PCR cuantitativa SYBR Green. Lo siguiente puede ayudar a los nuevos usuarios de instrumentos. Generalmente, el número de ciclos se representa gráficamente frente a la fluorescencia. Los ciclos umbral (CTs) o los puntos de cruce se utilizan para determinar la cantidad de molde en cada muestra. El ciclo umbral o punto de cruce es el primer ciclo que muestra un aumento detectable de la fluorescencia debido a la formación de productos de PCR. Los ciclos anteriores al punto de cruce son los ciclos de línea de base. Los ciclos de línea de base no muestran un aumento detectable de la fluorescencia debido a los productos de la PCR. El umbral utilizado para determinar cuándo se produce el primer aumento detectable de fluorescencia también puede ajustarse manualmente. El umbral debe hacerse siempre en un gráfico de amplificación logarítmica. En un gráfico de amplificación logarítmica, el umbral debe establecerse en el intervalo logarítmico-lineal y no en la fase de meseta.
Curvas de fusión
Realizar un análisis de curva de fusión al final del experimento ayudará a analizar únicamente el producto de PCR de interés. Siga las instrucciones del fabricante del instrumento de tiempo real para el análisis de la curva de fusión. Las ejecuciones sucesivas con los mismos cebadores pueden modificarse para eliminar la contribución de la formación de dímeros de cebadores a la señal del producto mediante la recopilación de datos en un paso cíclico adicional, cuya temperatura debe situarse entre las temperaturas de fusión del dímero y del producto ya determinadas (TMs).
Métodos de cuantificación
Curvas estándar
Las curvas estándar son necesarias tanto para la cuantificación absoluta como para la relativa. Al generar curvas estándar, deben utilizarse diferentes concentraciones de ADN (normalmente cinco) para generar una curva estándar que ponga entre paréntesis la concentración de la incógnita. Cada concentración debe realizarse por duplicado.
Cuantificación absoluta y relativa
Este kit SYBR Green PCR puede utilizarse para cuantificar el ADN diana mediante cuantificación absoluta o relativa. Las técnicas de cuantificación absoluta se utilizan para determinar la cantidad de ADN diana en la muestra inicial, mientras que la cuantificación relativa determina la relación entre la cantidad de ADN diana y un amplicón de referencia. El amplicón de referencia ideal tendría una expresión invariable y constitutiva. En la práctica, se elige un gen housekeeping para esta función, pero hay otras opciones de referencia que se ajustan mejor a los requisitos anteriores.1
La cuantificación absoluta utiliza estándares externos para determinar la cantidad absoluta de ácido nucleico diana de interés. Para eliminar las diferencias en la cuantificación debidas al recocido, los sitios de unión del cebador de los estándares externos deben ser los mismos que los de la secuencia diana. El estándar externo ideal contiene secuencias que son iguales a la secuencia diana o que sólo varían ligeramente de la secuencia diana. Para la cuantificación absoluta se necesitan eficiencias de amplificación equivalentes entre la diana y el estándar externo. Una vez identificado un constructo o amplicón adecuado, se genera una curva estándar de diluciones de estándar externo y se utiliza para determinar las concentraciones de muestras diana desconocidas.
La cuantificación relativa permite calcular la relación entre la cantidad de molde diana y un molde de referencia en una muestra. Dado que este método mide la cantidad de diana en relación con un control presumiblemente invariante, la qPCR relativa se utiliza con mayor frecuencia para medir diferencias de polimorfismo genético, por ejemplo, entre tejidos o entre muestras sanas y enfermas. La ventaja de esta técnica es que el uso de un patrón interno puede minimizar las variaciones en la preparación y manipulación de las muestras. Cuando se utilizan sistemas SYBR, la cuantificación de la diana y de la referencia interna deben realizarse en reacciones separadas.
La precisión de la cuantificación relativa depende de la elección adecuada de una plantilla de referencia para los estándares. La variabilidad del estándar influirá en los resultados, por lo que es muy importante que los estándares sean adecuados.1 Algunos investigadores optan por no ejecutar una curva estándar e informar de las cantidades objetivo como una fracción de la referencia, una técnica denominada cuantificación comparativa. Alternativamente, se puede asumir que las eficiencias de amplificación de la diana y la referencia son insignificantes y cuantificar la diana basándose únicamente en la curva estándar determinada para la secuencia de referencia. Por último, en la más precisa de las técnicas de cuantificación relativa, se miden las eficiencias de amplificación tanto de la referencia como de la diana y se determina un factor de corrección. Este proceso, denominado normalización,1 requiere una muestra que contenga concentraciones conocidas tanto de la diana como de la referencia y la generación de dos curvas estándar.
Determinación de las eficiencias de reacción de la PCR
La eficiencia de la PCR entre una muestra de referencia y una muestra diana se determina preparando una serie de diluciones para cada diana. Los valores de CT de la referencia se restan de la diana y esta diferencia de valores de CT se representa gráficamente frente al logaritmo de la cantidad de molde. Si la pendiente resultante de la línea recta es inferior a ± 0,1, se considera que las eficiencias de amplificación son similares.
Equipos
- Instrumento de PCR cuantitativa
- Microcentrífuga
- Campana de flujo laminar para el montaje de la PCR (opcional)
- Campana de flujo laminar para el montaje de la PCR (opcional)
- Campana de flujo laminar para el montaje de la PCR (opcional).
Suministros
- qPCR SYBR Green Mix - Consulte las guías de selección de qPCR (Parte 1 y Parte 2)
- Plantilla de ADNc/ADNc- reacción de ADNc diluida 1:10 para detectar dianas de media a alta expresión o 1:2 a 1:5 para transcritos poco frecuentes o de 10 ng a 100 ng de ADNg
- Incipientes de avance y retroceso diluidos hasta la concentración de trabajo (10µM de existencias de trabajo son suficientes para la mayoría de los ensayos)
- Preparaciones de ADNc para la detección de dianas de expresión media a alta.ul>
- También hay disponibles cebadores de expresión génica prediseñados para la mayoría de los organismos modelo (KiCqStart® SYBR® Green Primers, KSPQ12012)
Protocolo
Preparación
- Colocar todos los componentes de la reacción en hielo.
- Mezcle y, a continuación, centrifugue brevemente para recoger el contenido del fondo del tubo.
Reacción estándar con colorante SYBR Green I
Nota: Hemos observado que los ensayos realizados en KiCqStart ReadyMix son óptimos cuando se utiliza una concentración de cebador superior a la de la PCR convencional. En los protocolos siguientes, utilizamos una concentración final de 450 nM, que hemos observado que es la concentración óptima para varios ensayos independientes. Prepare suficiente master mix para realizar todas las muestras por duplicado.
a. Asegúrese de incluir controles negativos (NTC) sin plantilla por duplicado.
b. Seleccione la tabla adecuada a continuación en función del reactivo de qPCR seleccionado.
c. Calcule la cantidad de reactivos a mezclar. Añada un 10% de volumen para tener en cuenta el error de pipeteo
d. Mezcle bien, evitando burbujas.
Mezcla magistral para reactivos KiCqStart:
Master Mix para otras qPCR completas ReadyMixes:
Master Mix para otras qPCR completas ReadyMixes.
Master Mix para reactivos qPCR con componentes separados:
Master Mix para reactivos qPCR con componentes separados:
Master Mix para reactivos qPCR con componentes separados.
1La concentración óptima de MgCl2 puede oscilar entre 1mM y 6mM.
2Consulte Apéndice 1 para determinar la concentración óptima de colorante de referencia para su instrumento.
2. Prepare las reacciones:
a. Para reacciones NTC, añada 4 μL de agua al tubo de reacción.
b. Para las reacciones experimentales, añada 4 μL de solución de ADNc al tubo de reacción.
c. Centrifugue brevemente todos los tubos. Confirme visualmente que todos los tubos o pocillos contienen muestra en el fondo en el volumen correcto.
d. Alícuota cuidadosa de 16 μL de mezcla maestra de plantilla en cada tubo o pocillo de placa de qPCR.
e. Mezcle bien las reacciones y gírelas si es necesario.
f. Tapar los tubos o sellar la placa PCR y etiquetar (según los requisitos del instrumento). (Asegúrese de que el etiquetado no obstruya la trayectoria de la luz de excitación/detección del instrumento. 3. Ejecute las muestras según las recomendaciones del fabricante del instrumento. A continuación se incluyen ejemplos de ciclos estándar y rápidos.
Parámetros de ciclismo rápido:
Nota: Utilice el protocolo de curva de disociación estándar (recopilación de datos).
4. Utilice el protocolo de curva de disociación estándar (recopilación de datos). Consulte el manual del instrumento para obtener orientación sobre cómo analizar los datos.
Concentraciones de colorante de referencia recomendadas para su uso con instrumentos. Para reactivos qPCR con un colorante de referencia independiente
Guía de solución de problemas
Materiales
Para seguir leyendo, inicie sesión o cree una cuenta.
¿No tiene una cuenta?