Secuenciación Sanger Pasos y método
¿Qué es la secuenciación Sanger?
La secuenciación Sanger, también conocida como "método de terminación de cadena", es un método para determinar la secuencia de nucleótidos del ADN. El método fue desarrollado por el dos veces Premio Nobel Frederick Sanger y sus colegas en 1977, de ahí el nombre de Secuenciación Sanger.
Para revisar la estructura general del ADN, por favor vea Figura 2.
¿Cómo funciona la secuenciación Sanger?
La secuenciación Sanger puede realizarse de forma manual o, más comúnmente, de forma automatizada a través de una máquina secuenciadora (Figura 1). Cada método sigue tres pasos básicos, que se describen a continuación.
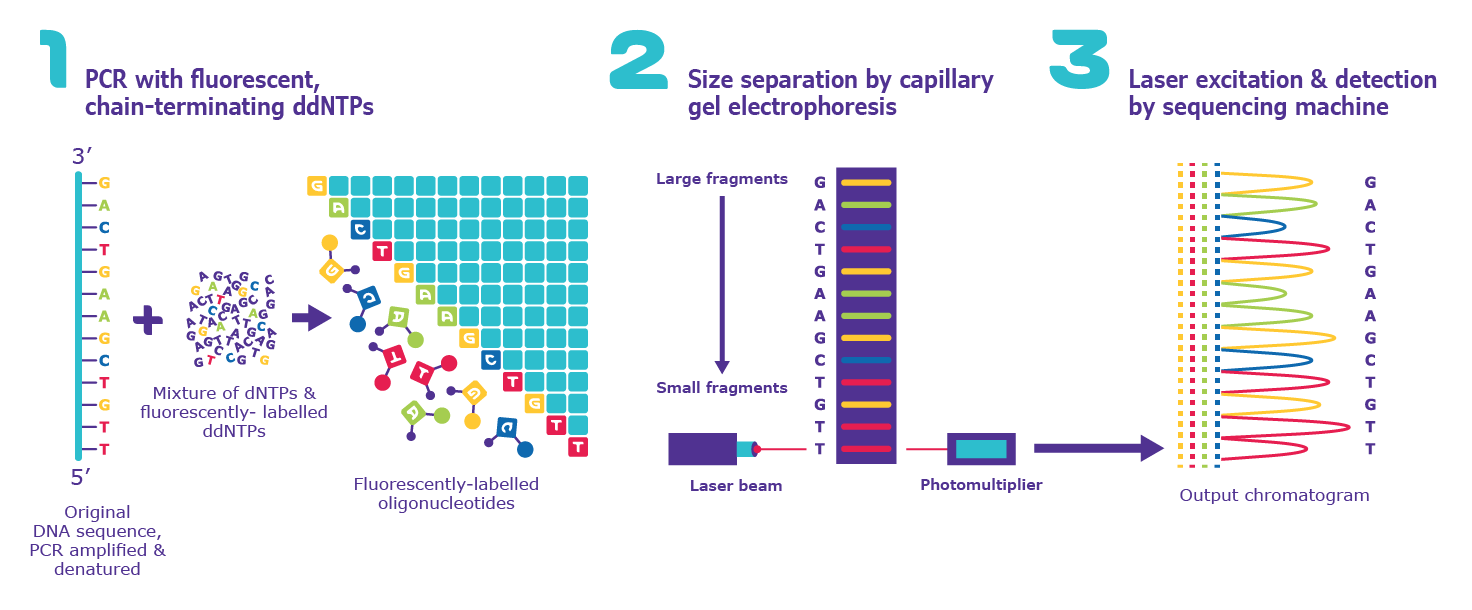
Figura 1.Tres pasos básicos de la secuenciación Sanger automatizada.
Pasos de la secuenciación Sanger
Hay tres pasos principales en la secuenciación Sanger.
1. Secuencia de ADN. Secuencia de ADN para la PCR de terminación de cadena
La secuencia de ADN de interés se utiliza como plantilla para un tipo especial de PCR llamada PCR de terminación en cadena. La PCR de terminación en cadena funciona igual que la PCR estándar, pero con una diferencia importante: la adición de nucleótidos modificados (dNTPs) llamados dideoxirribonucleótidos (ddNTPs). En el paso de extensión de la PCR estándar, la ADN polimerasa añade dNTPs a una cadena de ADN en crecimiento catalizando la formación de un enlace fosfodiéster entre el grupo 3'-OH libre del último nucleótido y el 5'-fosfato del siguiente (Figura 2).
En la PCR de terminación de cadena, el usuario mezcla una proporción baja de ddNTPs de terminación de cadena con los dNTPs normales en la reacción de PCR. Los ddNTPs carecen del grupo 3'-OH necesario para la formación del enlace fosfodiéster; por lo tanto, cuando la ADN polimerasa incorpora un ddNTP al azar, cesa la extensión. El resultado de la PCR con terminación en cadena son millones o miles de millones de copias de oligonucleótidos de la secuencia de ADN de interés, terminadas en una longitud aleatoria (n) por 5'-ddNTPs.
En la secuenciación Sanger manual, se preparan cuatro reacciones de PCR, cada una con un único tipo de ddNTP (ddATP, ddTTP, ddGTP y ddCTP).
En la secuenciación automatizada Sanger, todos los ddNTPs se mezclan en una sola reacción, y cada uno de los cuatro dNTPs tiene una etiqueta fluorescente única.
2. Separación por tamaño mediante electroforesis en gel
En el segundo paso, los oligonucleótidos terminados en cadena se separan por tamaño mediante electroforesis en gel. En la electroforesis en gel, las muestras de ADN se cargan en un extremo de una matriz de gel y se aplica una corriente eléctrica; el ADN tiene carga negativa, por lo que los oligonucleótidos serán atraídos hacia el electrodo positivo del lado opuesto del gel. Dado que todos los fragmentos de ADN tienen la misma carga por unidad de masa, la velocidad a la que se mueven los oligonucleótidos vendrá determinada únicamente por el tamaño. Cuanto más pequeño sea un fragmento, menos fricción experimentará al desplazarse por el gel y más rápido se moverá. Como resultado, los oligonucleótidos se ordenarán de menor a mayor, leyendo el gel de abajo a arriba.
En la secuenciación Sanger manual, los oligonucleótidos de cada una de las cuatro reacciones de PCR se corren en cuatro carriles separados de un gel. Esto permite al usuario saber qué oligonucleótidos corresponden a cada ddNTP.
En la secuenciación automatizada de Sanger, todos los oligonucleótidos se ejecutan en una única electroforesis en gel capilar dentro de la máquina de secuenciación.
3. Análisis del gel y determinación de la secuencia del ADN
El último paso consiste simplemente en leer el gel para determinar la secuencia del ADN de entrada. Debido a que la ADN polimerasa sólo sintetiza ADN en la dirección 5' a 3' a partir de un cebador proporcionado, cada ddNTP terminal corresponderá a un nucleótido específico en la secuencia original (por ejemplo, el fragmento más corto debe terminar en el primer nucleótido desde el extremo 5', el segundo fragmento más corto debe terminar en el segundo nucleótido desde el extremo 5', etc.) Por lo tanto, leyendo las bandas del gel de menor a mayor, podemos determinar la secuencia 5' a 3' de la cadena de ADN original.
En la secuenciación Sanger manual, el usuario lee los cuatro carriles del gel a la vez, de abajo a arriba, utilizando el carril para determinar la identidad del ddNTP terminal de cada banda. Por ejemplo, si la banda inferior se encuentra en la columna correspondiente a ddGTP, entonces el fragmento de PCR más pequeño termina con ddGTP, y el primer nucleótido desde el extremo 5' de la secuencia original tiene una base de guanina (G).
En la secuenciación automatizada Sanger, un ordenador lee cada banda del gel capilar, en orden, utilizando la fluorescencia para llamar la identidad de cada ddNTP terminal. En pocas palabras, un láser excita las etiquetas fluorescentes de cada banda y un ordenador detecta la luz emitida. Como cada uno de los cuatro ddNTPs está marcado con una etiqueta fluorescente diferente, la luz emitida puede relacionarse directamente con la identidad del ddNTP terminal. El resultado se denomina cromatograma, que muestra el pico fluorescente de cada nucleótido a lo largo del ADN molde.
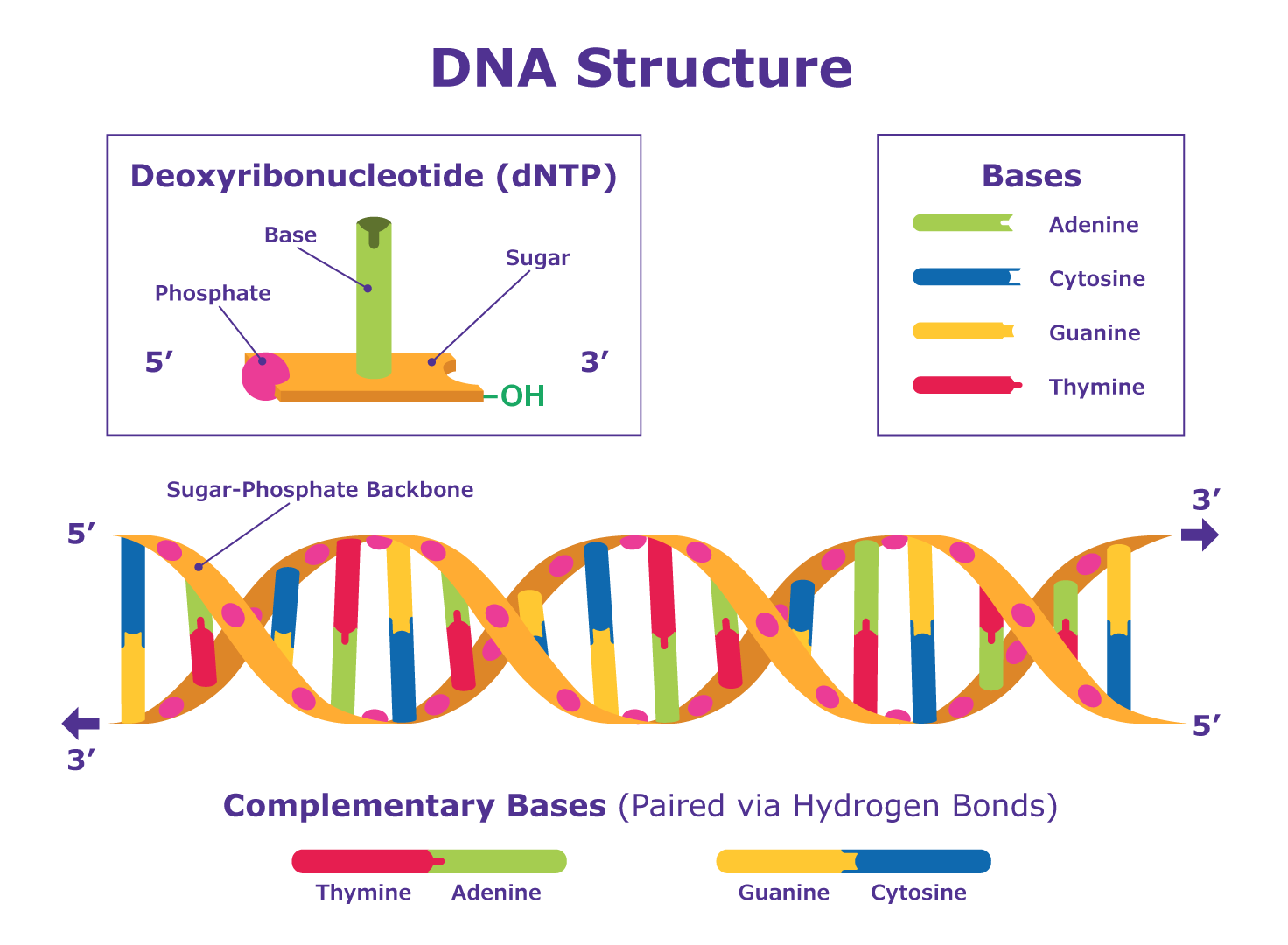
Figura 2.Esquema de la estructura del ADN. El ADN es una molécula compuesta por dos cadenas que se enrollan una alrededor de la otra para formar una doble hélice. Cada hebra está formada por una cadena de moléculas llamadas desoxirribonucleótidos (dNTPs).
Cada dNTP contiene un grupo fosfato, un grupo azúcar y una de las cuatro bases nitrogenadas [adenina (A), timina (T), guanina (G) o citosina (C)]. Los dNTPs se encadenan de forma lineal mediante enlaces covalentes fosfodiéster entre el azúcar de un dNTP y el grupo fosfato del siguiente; este patrón repetido de azúcar-fosfato constituye la espina dorsal de azúcar-fosfato.
Las bases nitrogenadas de las dos hebras separadas se unen mediante enlaces de hidrógeno entre bases complementarias para formar la hélice de ADN de doble cadena.
Cómo leer los resultados de la secuenciación Sanger
La lectura adecuada de los resultados de la secuenciación Sanger dependerá de cuál de las dos cadenas complementarias de ADN sea de interés y de qué cebador se disponga. Si las dos hebras de ADN son A y B y la hebra A es de interés, pero el cebador es mejor para la hebra B, los fragmentos de salida serán idénticos a la hebra A. Por otro lado, si la hebra A es de interés y el cebador es mejor para la hebra A, entonces la salida será idéntica a la hebra B. Por lo tanto, la salida debe ser convertida de nuevo a la cadena A.
Así, si la secuencia de interés es "TACG" y el cebador es mejor para esa cadena, la salida será "ATGC" y, por lo tanto, debe ser convertida de nuevo a "TACG". Sin embargo, si el cebador es mejor para la cadena complementaria ("ATGC"), entonces la salida será "TACG", que es la secuencia correcta.
En resumen, antes de empezar, ¡necesita saber cuál es su objetivo y cómo va a llegar a él! Así que teniendo esto en cuenta, aquí tienes un ejemplo del ejemplo anterior (TACG -> ATGC -> TACG). Si las etiquetas de los dideoxinucleótidos son T = amarillo, A = rosa, C = azul oscuro y G = azul claro, obtendrás las secuencias cortas primer-A, primer-AT, primer-ATG y primer-ATGC. Una vez separados los fragmentos por electroforesis, el láser leerá los fragmentos en orden de longitud (rosa, amarillo, azul claro y azul oscuro) y producirá un cromatograma. El ordenador convertirá las letras, por lo que la secuencia final es la correcta TACG.
Secuenciación Sanger vs. PCR
La secuenciación de Sanger y la PCR utilizan materiales de partida similares y pueden utilizarse conjuntamente, pero ninguna puede sustituir a la otra.
La PCR se utiliza para amplificar el ADN en su totalidad. Aunque pueden producirse fragmentos de longitudes variables por accidente (por ejemplo, la ADN polimerasa podría caerse), el objetivo es duplicar la secuencia completa de ADN. Para ello, los "ingredientes" son el ADN diana, los nucleótidos, el cebador de ADN y la ADN polimerasa (concretamente la Taq polimerasa, que puede sobrevivir a las altas temperaturas que requiere la PCR).
En cambio, el objetivo de la secuenciación Sanger es generar cada longitud posible de ADN hasta la longitud completa del ADN diana. Por eso, además de los materiales de partida de la PCR, son necesarios los dideoxinucleótidos.
La secuenciación Sanger y la PCR pueden unirse al generar el material de partida para un protocolo de secuenciación Sanger. La PCR se puede utilizar para crear muchas copias del ADN que se va a secuenciar.
Tener más de una plantilla a partir de la cual trabajar hace que el protocolo Sanger sea más eficiente. Si la secuencia diana tiene 1.000 nucleótidos de longitud y sólo hay una copia de la plantilla, va a llevar más tiempo generar los 1.000 fragmentos marcados. Sin embargo, si hay varias copias de la plantilla, en teoría se tardará menos tiempo en generar los 1.000 fragmentos marcados.
Productos relacionados
Para seguir leyendo, inicie sesión o cree una cuenta.
¿No tiene una cuenta?