Protocole universel de qPCR SYBR Green
Vue d'ensemble de la technologie
Considérations relatives à l'essai
Méthodes de quantification
Equipement & ; Supplies
Guide de sélection des mélanges pour PCR
Guide de sélection des mélanges pour PCR
.br> Protocol
Troubleshooting
Materials
Références
Vue d'ensemble de la technologie : SYBR Green qPCR
Avec le développement de thermocycleurs incorporant une détection fluorescente, la PCR a une nouvelle application innovante. Dans la PCR de routine, le résultat critique est la quantité finale d'amplicon générée après le processus. La PCR en temps réel ou quantitative et la RT-PCR utilisent la linéarité de l'amplification de l'ADN pour déterminer les quantités absolues ou relatives d'une séquence connue dans un échantillon. En utilisant un rapporteur fluorescent dans la réaction, il est possible de mesurer la génération d'ADN.
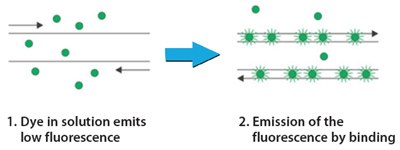
Figure 1.Aperçu de la technologie : SYBR Green qPCR
A) Les différentes phases de la réaction:
Base: La concentration initiale de la matrice est faible ; par conséquent, l'intensité de la fluorescence est trop faible pour être détectée et seul le signal de fond est évident.
Exponentiel: Après que le rendement de la cible a atteint le seuil de détection, représenté par la ligne de seuil rouge, le cours de la réaction peut être suivi à travers la phase exponentielle.
Linéaire: Au fur et à mesure que la concentration de la matrice augmente, la concentration d'ADN polymérase disponible diminue et la vitesse de réaction diminue.
Plateau: Il n'y a pas assez d'enzyme libre pour continuer l'amplification et donc, après ce point, la réaction est au rendement maximum, ou la phase de plateau.
B) Les réactions individuelles sont caractérisées par le cycle au cours duquel la fluorescence dépasse pour la première fois le seuil, appelé cycle de quantification (Cq). Si le matériel de départ est abondant, l'amplification est observée dans les premiers cycles et le Cq est plus faible. Si le matériel de départ est rare, l'amplification est observée dans les cycles plus tardifs et la Cq est plus élevée. Cette corrélation entre la fluorescence, Cq, et la quantité de produit amplifié permet de quantifier la matrice sur une large plage dynamique.
La PCR en temps réel se prête également aux études relatives. Une réaction peut être effectuée à l'aide d'amorces propres à chaque région à amplifier et marquées avec différents colorants fluorescents. Plusieurs thermocycleurs quantitatifs disponibles dans le commerce comportent plusieurs canaux de détection. Dans ce système multiplex, la quantité d'ADN/ADN cible peut être comparée à la quantité d'une séquence d'entretien, par exemple la GAPDH ou la β-actine.
NR= non recommandé
X= recommandé
XX= méthode préférée
NR= non recommandé
X= recommandé
XX= méthode préférée
Méthode préférée
Considérations relatives à l'essai
Préparation de l'ADN
L'étape la plus importante pour garantir le succès de la PCR est une préparation de l'ADN de haute qualité. L'intégrité et la pureté de la matrice d'ADN sont essentielles. La PCR quantitative implique plusieurs cycles de réactions enzymatiques et est donc plus sensible aux impuretés telles que les protéines, le phénol/chloroforme, les sels, l'EDTA et d'autres solvants chimiques. Les contaminants peuvent également interférer avec la détection de la fluorescence. Le rapport des valeurs d'absorbance à 260 nM et 280 nM donne une estimation de la pureté de l'ADN. L'ADN pur présente un rapport A260/A280 de 1,8 à 2,0. Des rapports inférieurs indiquent la présence de contaminants tels que des protéines.
Template
Très peu de copies d'acide nucléique cible (équivalant à environ 100 pg d'ADNg ou d'ADNc) sont nécessaires pour initier la qPCR. Pour minimiser la contamination par des inhibiteurs de réaction, la quantité de matrice de départ doit être maintenue au minimum requis pour obtenir une quantification précise. Lorsque le matériel de départ est de l'ARN, la conception d'amorces et le traitement à la DNase I réduiront les signaux qui peuvent être générés par la contamination de l'ADNg.
Conception d'amorces
Que l'on utilise un colorant se liant à l'ADNdb ou une chimie de détection basée sur une sonde, la conception d'amorces de haute qualité est l'une des étapes pré-expérimentales les plus cruciales de la qPCR. Les amorces spécifiques pour la PCR doivent être conçues à l'aide d'un logiciel de conception d'amorces afin d'éliminer les complications introduites par les diluants d'amorces et les structures secondaires. Des concentrations d'amorces plus faibles diminuent l'accumulation de la formation de dimères d'amorces et la formation de produits non spécifiques, ce qui est essentiel lors de l'utilisation du colorant SYBR Green I dans la PCR quantitative.
dNTPs
Les mélanges maîtres PCR/qPCR standard contiennent du dATP, du dCTP, du dGTP et du dTTP. Toutefois, il existe des mélanges qui remplacent le dTTP par du dUTP. Les produits des réactions précédentes effectuées avec du dUTP contiendront de l'uracile au lieu de la thymine. Ils sont alors susceptibles d'être clivés par l'Uracil-DNA-Glycosylase (UNG). Par conséquent, l'incubation préalable des réactions suivantes avec l'UNG empêche la contamination entre les réactions. Pour être efficaces, toutes les réactions du laboratoire doivent utiliser du dUTP.
Concentration de magnésium
Le chlorure de magnésium (MgCl2) est nécessaire à la transcriptase inverse, à l'ADN polymérase Taq et à l'activité de l'exonucléase 5' à 3' de l'ADN Taq. Les concentrations optimales de Mg2+ pour les réactions contenant de la DLP sont généralement comprises entre 3 et 6 mM. Des concentrations plus faibles de chlorure de magnésium entraînent généralement la formation de moins de produits non spécifiques. Certaines solutions ReadyMix sont fournies à une concentration 2X de 7 mM de chlorure de magnésium (concentration finale de 3,5 mM). Dans certains cas, un flacon d'une solution de chlorure de magnésium 25 mM est fourni pour optimiser davantage la concentration finale de chlorure de magnésium si nécessaire. Un mélange réactionnel ne contenant pas de MgCl2 peut parfois être nécessaire afin de pouvoir utiliser une faible concentration, par exemple lors de l'utilisation de la détection par sonde Scorpion.
Transcriptase inverse
Une enzyme de transcriptase inverse qui fournit des rendements élevés d'ADNc, tout en conservant son activité à haute température, est essentielle au succès de la RT-qPCR. La performance à haute température permet de s'assurer que les régions de l'ARN ayant une structure secondaire significative sont déstabilisées et accessibles pour l'hybridation et l'amplification subséquente. Lors de la RT-qPCR en une étape, la performance à haute température permet l'utilisation d'amorces spécifiques du gène avec des températures de fusion élevées (Tm), ce qui augmente la spécificité de la réaction. Lors de l'exécution de protocoles en deux étapes, il est important de veiller à ce que l'enzyme produise un rendement linéaire et proportionnel d'ADNc à partir d'ARN. La réduction du pipetage peut diminuer la variabilité. Certains ReadyMix contiennent des amorces et d'autres réactifs nécessaires à la réalisation de la RT, par exemple le ReadyScript® cDNA Synthesis Mix (RDRT).
Taq DNA Polymerase
De même que la sélection de la transcriptase inverse la plus appropriée pour la RT, la sélection de l'enzyme appropriée est vitale. Un problème fondamental de l'ADN polymérase Taq naturelle est que l'enzyme a une activité résiduelle à basse température. La liaison non spécifique de l'amorce entraîne la formation d'un produit non spécifique en raison de cette activité résiduelle de la polymérase. Les ADN polymérases Taq bloquées par des anticorps ou des produits chimiques ("démarrage à chaud") permettent de remédier à cette situation en empêchant l'activité de l'enzyme jusqu'à ce que l'étape de dénaturation à haute température commence. Reportez-vous au Guide de sélection des mélanges PCR pour définir la meilleure polymérase à démarrage rapide pour votre application.
Colorant de référence interne par type d'instrument
Certains thermocycleurs de PCR en temps réel nécessitent un colorant de chargement tel que le ROX pour contrôler la variabilité du système optique et pour normaliser les différences d'intensité du signal. De même, certains thermocycleurs nécessitent de la fluorescéine pour créer un fond virtuel lorsqu'on travaille avec des essais au colorant SYBR Green I (dont le fond est très faible). Ces éléments peuvent être fournis dans le ReadyMix ou en tant que composants séparés afin que la concentration appropriée puisse être utilisée. Dans certains cas, un flacon de colorant de référence interne est inclus pour la normalisation de la réaction. L'excitation maximale de ce colorant est de 586 nM et l'émission maximale est de 605 nM. Les réglages standard de l'instrument pour le colorant de référence ROX sont satisfaisants pour la mesure du colorant de référence interne. Ce colorant de référence interne est nécessaire pour les systèmes de détection de séquences ABI.
Instruments
Les réactifs compatibles avec les instruments devront être sélectionnés. Les plates-formes utilisent des colorants de normalisation différents ; il convient donc de sélectionner des réactifs avec des colorants de normalisation compatibles (voir Appendix 1).q = 2 fois sur 96 puits de réplication) et un système optique qui excite et détecte l'émission de manière aussi sensible et uniforme que possible sur une large gamme de longueurs d'onde. Cela permet un large choix de fluorophores et un multiplexage. D'autres caractéristiques à prendre en compte sont les coûts d'exploitation associés à des consommables spécifiques, par exemple si une plaque de microtitration standard n'est pas utilisée pour les réactions, ainsi que la commodité de chargement des plaques/tubes de format non standard.
Contrôles
Un contrôle positif est toujours utile pour s'assurer que tous les composants de la trousse fonctionnent correctement. Un contrôle sans modèle/négatif est nécessaire pour déterminer s'il y a contamination. Un signal dans le contrôle sans modèle démontre la présence d'une contamination par l'ADN ou la formation de dimères d'amorces.
Buffer
Les tampons ou les mélanges maîtres de réaction contiennent généralement des dNTP, une Taq ADN polymérase, du MgCl2, et des stabilisateurs. Le SYBR Green I, le ROX™, la fluorescéine et des colorants de chargement inertes peuvent également être inclus, en fonction des exigences de la chimie de détection, de l'instrument et de la réaction. Les composants du tampon PCR et les stabilisateurs sont généralement la propriété du fabricant. S'ils sont achetés séparément, une flexibilité maximale est possible, puisque chaque ingrédient peut être optimisé individuellement dans la réaction. En revanche, si l'achat des ingrédients ensemble sous forme de mastermix réduit la flexibilité, il augmente la cohérence et la commodité des lots tout en réduisant le nombre d'étapes de pipetage et, par conséquent, les risques d'erreur et de contamination.
Analyse des données
Suivez les recommandations de l'instrument en temps réel utilisé pour effectuer la PCR SYBR Green quantitative. Ce qui suit peut aider les nouveaux utilisateurs d'instruments. En général, le nombre de cycles est représenté en fonction de la fluorescence. Les cycles seuils (CT) ou les points de passage sont utilisés pour déterminer la quantité de matrice dans chaque échantillon. Le cycle seuil ou point de passage est le premier cycle qui montre une augmentation détectable de la fluorescence due à la formation de produits PCR. Les cycles précédant le point de croisement sont les cycles de base. Les cycles de base ne montrent pas d'augmentation détectable de la fluorescence due aux produits PCR. Le seuil utilisé pour déterminer le moment où se produit la première augmentation détectable de la fluorescence peut également être ajusté manuellement. Le seuil doit toujours être déterminé sur un diagramme d'amplification logarithmique. Dans un diagramme d'amplification logarithmique, le seuil doit être fixé dans la plage log-linéaire et non dans la phase de plateau.
Courbes de fusion
La réalisation d'une analyse de la courbe de fusion à la fin de la série permettra d'analyser uniquement le produit PCR d'intérêt. Suivre les instructions du fabricant de l'instrument en temps réel pour l'analyse de la courbe de fusion. Les passages successifs avec les mêmes amorces peuvent être modifiés pour éliminer la contribution de la formation de dimères d'amorces au signal du produit en collectant des données lors d'une étape de cyclage supplémentaire, dont la température doit se situer entre les températures de fusion du dimère et du produit déjà déterminées (TMs).
Méthodes de quantification
Courbes standard
Les courbes standard sont nécessaires pour la quantification absolue et relative. Lors de la création de courbes standard, différentes concentrations d'ADN (généralement cinq) doivent être utilisées pour créer une courbe standard qui correspondra à la concentration de l'inconnu. Chaque concentration doit être exécutée en double.
Quantification absolue et relative
Ce kit PCR SYBR Green peut être utilisé pour quantifier l'ADN cible en utilisant soit la quantification absolue, soit la quantification relative. Les techniques de quantification absolue sont utilisées pour déterminer la quantité d'ADN cible dans l'échantillon initial, tandis que la quantification relative détermine le rapport entre la quantité d'ADN cible et un amplicon de référence. L'amplicon de référence idéal devrait avoir une expression invariable et constitutive. Dans la pratique, un gène de ménage est choisi pour cette fonction, mais il existe d'autres choix de référence qui répondent mieux aux exigences susmentionnées.1
La quantification absolue utilise des étalons externes pour déterminer la quantité absolue de l'acide nucléique cible d'intérêt. Pour éliminer les différences de quantification dues au recuit, les sites de liaison des amorces des étalons externes doivent être les mêmes que ceux de la séquence cible. L'étalon externe idéal contient des séquences qui sont identiques à la séquence cible ou qui ne varient que légèrement par rapport à la séquence cible. Des efficacités d'amplification équivalentes entre la cible et l'étalon externe sont nécessaires pour une quantification absolue. Une fois qu'une construction ou un amplicon approprié est identifié, une courbe standard de dilutions de l'étalon externe est générée et utilisée pour déterminer les concentrations d'échantillons cibles inconnus.
La quantification relative permet de calculer le rapport entre la quantité de matrice cible et une matrice de référence dans un échantillon. Étant donné que cette méthode mesure la quantité de cible par rapport à un contrôle supposé invariant, la qPCR relative est le plus souvent utilisée pour mesurer les différences de polymorphisme génétique, par exemple, entre les tissus ou entre les échantillons sains et malades. L'avantage de cette technique est que l'utilisation d'un étalon interne permet de minimiser les variations dans la préparation et la manipulation des échantillons. Lors de l'utilisation de systèmes SYBR, la quantification de la cible et de la référence interne doit être effectuée dans des réactions séparées.
La précision de la quantification relative dépend du choix approprié d'un modèle de référence pour les étalons. La variabilité de l'étalon influencera les résultats et il est donc très important que les étalons soient appropriés.1 Certains chercheurs choisissent de ne pas exécuter de courbe d'étalonnage et de rapporter les quantités de la cible comme une fraction de la référence, une technique appelée quantification comparative. Il est également possible de supposer que les efficacités d'amplification de la cible et de la référence sont négligeables et de quantifier la cible en se basant uniquement sur la courbe standard déterminée pour la séquence de référence. Enfin, dans la plus précise des techniques de quantification relative, les efficacités d'amplification de la référence et de la cible sont mesurées et un facteur de correction est déterminé. Ce processus, appelé normalisation,1 nécessite un échantillon contenant des concentrations connues de la cible et de la référence et la génération de deux courbes standard.
Détermination des efficacités de réaction PCR
L'efficacité de la PCR entre un échantillon de référence et un échantillon cible est déterminée en préparant une série de dilutions pour chaque cible. Les valeurs CT de la référence sont soustraites de la cible et cette différence de valeurs CT est tracée en fonction du logarithme de la quantité de matrice. Si la pente de la ligne droite qui en résulte est inférieure à ± 0,1, les efficacités d'amplification sont jugées similaires.
Equipement<
- Instrument de PCR quantitative
- Microcentrifugeuse
- Hotte à flux laminaire pour la mise en place de la PCR (en option)
Fournitures
- qPCR SYBR Green Mix - Reportez-vous aux guides de sélection qPCR (Part 1 et Part 2)
- Modèle ADN/ADN - réaction ADNc diluée à 1 :10 pour détecter des cibles moyennement ou fortement exprimées ou 1:2 à 1 :5 pour les transcrits rares ou 10 ng à 100 ng d'ADNg
- Les amorces directes et inverses sont diluées à la concentration de travail (des stocks de travail de 10µM sont suffisants pour la plupart des essais)
- La réaction ADN/ADN est diluée à la concentration de travail.ul>
- Des amorces d'expression génique prédéfinies sont également disponibles pour la plupart des organismes modèles (KiCqStart® SYBR® ;Green Primers, KSPQ12012)
Protocole
Préparation
- Placez tous les composants de la réaction sur de la glace.
- Mélanger puis centrifuger brièvement pour recueillir le contenu au fond du tube.
Réaction standard au colorant SYBR Green I
Note: Nous avons observé que les essais réalisés dans KiCqStart ReadyMix sont optimaux lorsque l'on utilise une concentration d'amorces plus élevée que dans la PCR conventionnelle. Dans les protocoles ci-dessous, nous utilisons une concentration finale de 450 nM, ce qui s'est avéré être la concentration optimale pour plusieurs essais indépendants.
1. Préparer suffisamment de mélange maître pour exécuter tous les échantillons en double.
a. Veillez à inclure des contrôles négatifs sans modèle en double.
b. Sélectionner le tableau approprié ci-dessous en fonction du réactif qPCR choisi.
c. Calculer la quantité de réactifs à mélanger. Ajouter 10% de volume pour tenir compte des erreurs de pipetage
d. Bien mélanger, en évitant les bulles.
Master Mix pour les réactifs KiCqStart:
La quantité de réactifs à mélanger doit être calculée en tenant compte de l'erreur de pipetage
d.
Master Mix for Other Complete qPCR ReadyMixes:
Mélange principal pour d'autres mélanges prêts pour la qPCR
Master Mix pour réactifs qPCR avec composants séparés:
Mélange de base pour réactifs qPCR avec composants séparés
1La concentration optimale de MgCl2 peut varier de 1mM à 6mM.
2 Consulter Appendix 1 pour déterminer la concentration optimale de colorant de référence pour votre instrument.
2. Préparer les réactions:
a. Pour les réactions NTC, ajoutez 4 μL d'eau au tube de réaction.
b. Pour les réactions expérimentales, ajouter 4 μL de solution d'ADNc au tube de réaction.
c. Centrifuger brièvement tous les tubes. Confirmer visuellement que tous les tubes ou puits contiennent l'échantillon au fond au volume correct.
d. Aliquotez soigneusement 16 μL de mélange maître de gabarit dans chaque tube qPCR ou puits de plaque.
e. Mélangez bien les réactions et faites tourner si nécessaire.
f. Boucher les tubes ou sceller la plaque PCR et l'étiqueter (selon les exigences de l'instrument). (Assurez-vous que l'étiquetage ne masque pas le trajet de la lumière d'excitation/détection de l'instrument.)
3. Exécutez les échantillons conformément aux recommandations du fabricant de l'instrument. Des exemples de cycles standard et rapide sont inclus ci-dessous.
Paramètres de cycle standard:
Paramètres de cyclage rapide : Les paramètres de cyclage rapide sont les suivants
Note: Utiliser le protocole standard de courbe de dissociation (collecte de données).
4. Consulter le manuel de l'instrument pour savoir comment analyser les données.
Appendice 1
Concentrations de colorant de référence recommandées pour l'utilisation avec les instruments. Pour les réactifs qPCR avec un colorant de référence séparé
Les concentrations de colorant de référence recommandées pour l'utilisation avec les instruments.
Guide de dépannage
Matériaux
Pour continuer à lire, veuillez vous connecter à votre compte ou en créer un.
Vous n'avez pas de compte ?