Séquençage Sanger : étapes et méthode
Qu'est-ce que le séquençage Sanger ?
Le séquençage Sanger, également connu sous le nom de "méthode de terminaison de chaîne", est une méthode permettant de déterminer la séquence de nucléotides de l'ADN. La méthode a été mise au point par Frederick Sanger, lauréat du prix Nobel, et ses collègues en 1977, d'où le nom de "séquence de Sanger".
Pour passer en revue la structure générale de l'ADN, veuillez vous reporter à la figure 2.
Comment fonctionne le séquençage Sanger ?
Le séquençage Sanger peut être effectué manuellement ou, plus couramment, de manière automatisée via une machine de séquençage (Figure 1). Chaque méthode suit trois étapes de base, décrites ci-dessous.
Séquençage Sanger
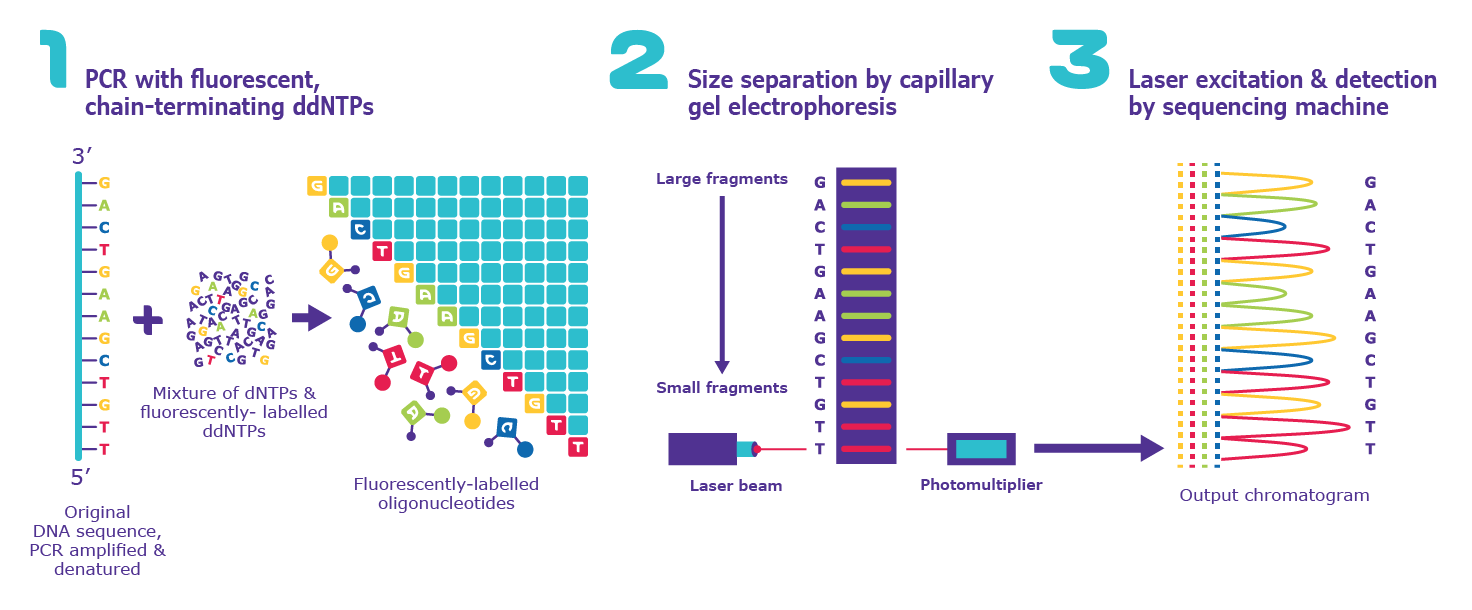
Figure 1.Trois étapes de base du séquençage automatisé de Sanger.
Etapes du séquençage Sanger
Le séquençage Sanger comporte trois étapes principales.
1. Séquence d'ADN pour la PCR de terminaison de chaîne
La séquence d'ADN d'intérêt est utilisée comme modèle pour un type spécial de PCR appelée PCR à terminaison en chaîne. La PCR à terminaison de chaîne fonctionne comme la PCR standard, mais avec une différence majeure : l'ajout de nucléotides modifiés (dNTP) appelés didésoxyribonucléotides (ddNTP). Lors de l'étape d'extension de la PCR standard, l'ADN polymérase ajoute des dNTP à un brin d'ADN en croissance en catalysant la formation d'une liaison phosphodiester entre le groupe 3'-OH libre du dernier nucléotide et le 5'-phosphate du suivant (Figure 2).
Dans la PCR à terminaison de chaîne, l'utilisateur mélange un faible rapport de ddNTP à terminaison de chaîne avec les dNTP normaux dans la réaction PCR. Les ddNTP ne possèdent pas le groupe 3'-OH nécessaire à la formation de la liaison phosphodiester par conséquent, lorsque l'ADN polymérase incorpore un ddNTP de manière aléatoire, l'extension s'arrête. Le résultat de la PCR avec terminaison en chaîne est constitué de millions ou de milliards de copies d'oligonucléotides de la séquence d'ADN concernée, terminées à des longueurs aléatoires (n) par des 5'-ddNTP.
Lors d'un séquençage Sanger manuel, quatre réactions PCR sont mises en place, chacune avec un seul type de ddNTP (ddATP, ddTTP, ddGTP et ddCTP) mélangé.
Dans le séquençage Sanger automatisé, tous les ddNTP sont mélangés dans une seule réaction, et chacun des quatre dNTP possède une étiquette fluorescente unique.
2 Séparation de taille par électrophorèse sur gel
Dans la deuxième étape, les oligonucléotides terminés par une chaîne sont séparés en fonction de leur taille par électrophorèse sur gel. Dans l'électrophorèse sur gel, les échantillons d'ADN sont chargés à une extrémité d'une matrice de gel et un courant électrique est appliqué ; l'ADN étant chargé négativement, les oligonucléotides sont attirés vers l'électrode positive située de l'autre côté du gel. Comme tous les fragments d'ADN ont la même charge par unité de masse, la vitesse à laquelle les oligonucléotides se déplacent n'est déterminée que par leur taille. Plus un fragment est petit, moins il subira de friction lors de son déplacement dans le gel, et plus il se déplacera rapidement. En conséquence, les oligonucléotides seront disposés du plus petit au plus grand, en lisant le gel de bas en haut.
Dans le séquençage Sanger&bsp;manuel les oligonucléotides de chacune des quatre réactions PCR sont exécutés dans quatre voies séparées d'un gel. Cela permet à l'utilisateur de savoir quels oligonucléotides correspondent à chaque ddNTP.
En séquençage Sanger automatisé tous les oligonucléotides sont exécutés dans une seule électrophorèse sur gel capillaire au sein de l'appareil de séquençage.
3 Analyse du gel et détermination de la séquence de l'ADN
La dernière étape consiste simplement à lire le gel pour déterminer la séquence de l'ADN d'entrée. Étant donné que l'ADN polymérase ne synthétise l'ADN que dans le sens 5' à 3' à partir d'une amorce fournie, chaque ddNTP terminal correspondra à un nucléotide spécifique dans la séquence d'origine (par exemple, le fragment le plus court doit se terminer à l'extrémité de l'extrémité de l'amorce), le fragment le plus court doit se terminer au premier nucléotide de l'extrémité 5', le deuxième fragment le plus court doit se terminer au deuxième nucléotide de l'extrémité 5', etc.) Par conséquent, en lisant les bandes du gel de la plus petite à la plus grande, nous pouvons déterminer la séquence 5' à 3' du brin d'ADN d'origine.
Dans le séquençage Sanger manuel, l'utilisateur lit les quatre bandes du gel en une seule fois, de bas en haut, en utilisant la bande pour déterminer l'identité du ddNTP terminal pour chaque bande. Par exemple, si la bande inférieure se trouve dans la colonne correspondant au ddGTP, le plus petit fragment PCR se termine par un ddGTP et le premier nucléotide à partir de l'extrémité 5' de la séquence originale a une base guanine (G).
Dans le séquençage Sanger automatisé, un ordinateur lit chaque bande du gel capillaire, dans l'ordre, en utilisant la fluorescence pour déterminer l'identité de chaque ddNTP terminal. En bref, un laser excite les marqueurs fluorescents de chaque bande et un ordinateur détecte la lumière émise. Comme chacun des quatre ddNTP est marqué par une étiquette fluorescente différente, la lumière émise peut être directement liée à l'identité du ddNTP terminal. Le résultat est appelé chromatogramme, qui montre le pic fluorescent de chaque nucléotide sur la longueur de l'ADN modèle.
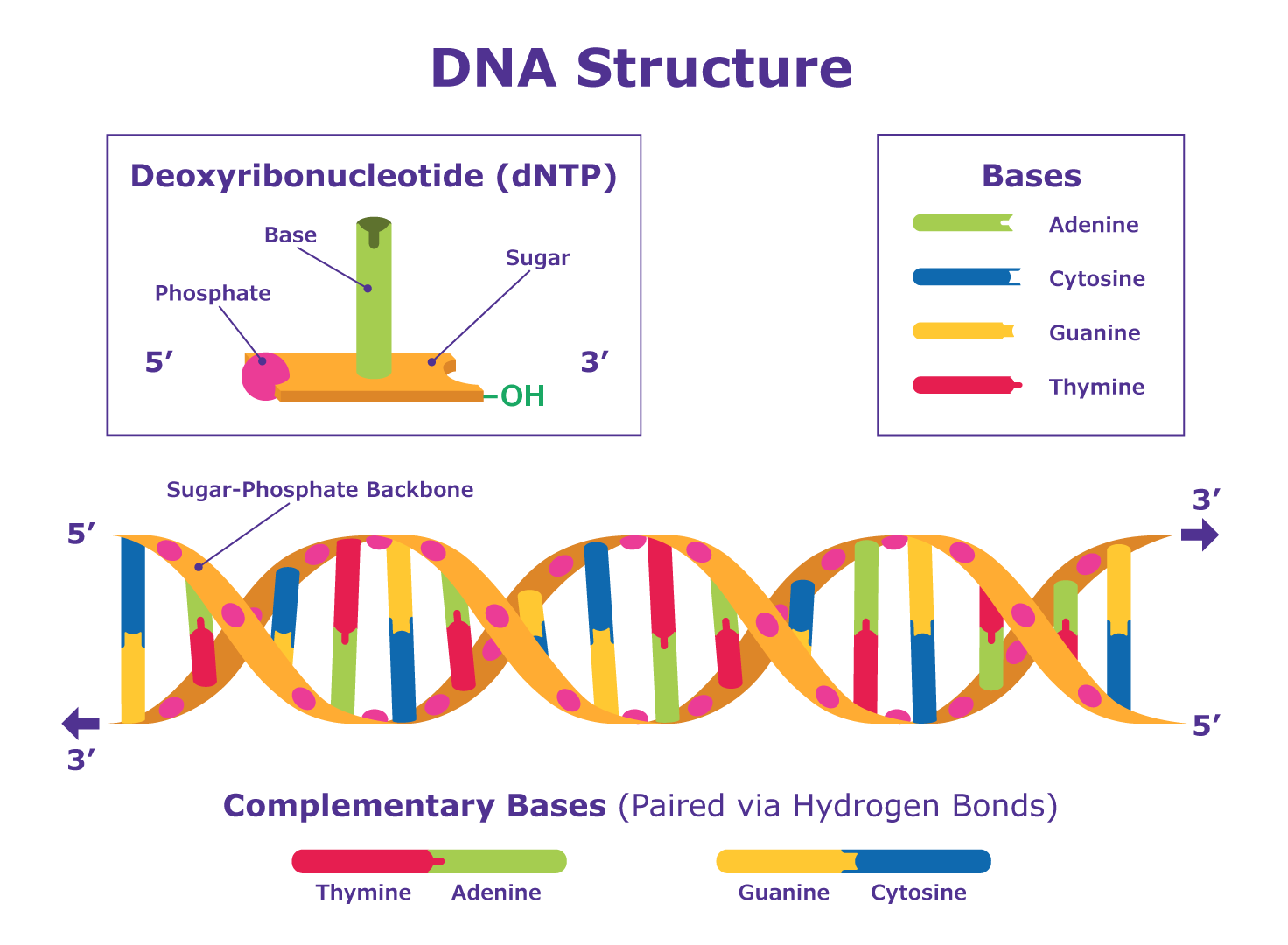
Figure 2.Schéma de la structure de l'ADN. L'ADN est une molécule composée de deux brins qui s'enroulent l'un autour de l'autre pour former une double hélice. Chaque brin est constitué d'une chaîne de molécules appelées désoxyribonucléotides (dNTP).
Les bases azotées des deux brins distincts sont liées par des liaisons hydrogène entre bases complémentaires pour former l'hélice double brin de l'ADN.
Comment lire les résultats du séquençage Sanger
La lecture correcte des résultats du séquençage Sanger dépendra de celui des deux brins d'ADN complémentaires qui est intéressant et de l'amorce disponible. Si les deux brins d'ADN sont A et B et que le brin A est intéressant, mais que l'amorce est meilleure pour le brin B, les fragments de sortie seront identiques au brin A. En revanche, si le brin A est intéressant et que l'amorce est meilleure pour le brin A, la sortie sera identique au brin B. Par conséquent, la sortie doit être reconvertie en brin A.
Ainsi, si la séquence d'intérêt se lit "TACG" et que l'amorce est meilleure pour ce brin, la sortie sera "ATGC" et, par conséquent, doit être reconvertie en "TACG". En revanche, si l'amorce est meilleure pour le brin complémentaire ("ATGC"), le résultat sera "TACG", qui est la séquence correcte.
En bref, avant de commencer, vous devez savoir ce que vous visez et comment vous allez y arriver ! En gardant cela à l'esprit, voici un exemple du premier exemple (TACG -> ; ATGC -> ; TACG). Si les étiquettes des didésoxynucléotides sont T = jaune, A = rose, C = bleu foncé et G = bleu clair, vous obtiendrez les courtes séquences suivantes : primer-A, primer-AT, primer-ATG et primer-ATGC. Une fois les fragments séparés par électrophorèse, le laser lit les fragments par ordre de longueur (rose, jaune, bleu clair et bleu foncé) et produit un chromatogramme. L'ordinateur convertit les lettres, de sorte que la séquence finale est le TACG correct.
Séquençage Sanger vs. PCR
Le séquençage Sanger et la PCR utilisent des matériaux de départ similaires et peuvent être utilisés conjointement, mais aucun ne peut remplacer l'autre.
La PCR est utilisée pour amplifier l'ADN dans son intégralité. Bien que des fragments de différentes longueurs puissent être produits par accident (par exemple, l'ADN polymérase peut se détacher), l'objectif est de dupliquer la séquence d'ADN dans son intégralité. À cette fin, les "ingrédients" sont l'ADN cible, les nucléotides, l'amorce d'ADN et l'ADN polymérase (en particulier la Taq polymérase, qui peut survivre aux températures élevées requises pour la PCR).
En revanche, l'objectif du séquençage Sanger est de générer toutes les longueurs d'ADN possibles jusqu'à la longueur totale de l'ADN cible. C'est pourquoi, en plus du matériel de départ de la PCR, les didésoxynucléotides sont nécessaires.
Le séquençage Sanger et la PCR peuvent être rapprochés lors de la génération du matériel de départ d'un protocole de séquençage Sanger. La PCR peut être utilisée pour créer de nombreuses copies de l'ADN qui doit être séquencé.
Le fait d'avoir plus d'une matrice à partir de laquelle travailler rend le protocole Sanger plus efficace. Si la séquence cible est longue de 1 000 nucléotides et qu'il n'y a qu'une seule copie de la matrice, il faudra plus de temps pour générer les 1 000 fragments marqués. En revanche, s'il y a plusieurs copies du modèle, il faudra théoriquement moins de temps pour générer les 1 000 fragments marqués.
Produits apparentés
Pour continuer à lire, veuillez vous connecter à votre compte ou en créer un.
Vous n'avez pas de compte ?