Analyse des données PCR/qPCR
- Analyse qualitative de la PCR/qPCR
- Analyse des données de la PCR/qPCR
- L'analyse des données de la PCR/qPCRDérivation de valeurs Cq précises
- Réglage du seuil
- qpcrq
- Stratégies de quantification par qPCR
- Quantification des courbes standard
- Quantification relative/comparative
- Normalisation
- Sélection des gènes de référence
- Analyse de la stabilité des gènes de référence
- Méthodes de normalisation alternatives
- Analyse statistique et visualisation des données
- Techniques de visualisation pour l'analyse univariée
- Tests statistiques
- Clustering hiérarchique
- Analyse en composantes principales
PCR/qPCR Analyse qualitative des données
Après une PCR traditionnelle, les données sont analysées par résolution sur un gel d'agarose ou, plus récemment, sur un système d'électrophorèse capillaire. Pour certaines applications, une qPCR sera effectuée avec les données de point final utilisées pour l'analyse, comme pour le génotypage SNP. Dans tous les cas, les données de point final fournissent une analyse qualitative après que la PCR a atteint une phase de plateau. Dans certains cas, il peut être possible d'analyser les données de point final pour réaliser une analyse semi-quantitative du rendement de la PCR, mais les mesures quantitatives sont plus souvent réalisées à l'aide de la qPCR et de l'analyse des valeurs du cycle de quantification (Cq)1 valeurs.
qPCR Data Analysis
Tout au long de ce guide, les facteurs qui contribuent aux variations de la mesure de l'acide nucléique par PCR ou qPCR ont été mis en évidence. Chacun de ces facteurs doit être optimisé pour aboutir à un essai qui fournit la valeur la plus proche possible de la quantité réelle de gène (cible) dans la réaction. Le résultat de ces processus est la génération d'un ensemble de valeurs Cq ; pour chaque cible dans chaque échantillon. Le processus de dérivation et d'analyse de ces valeurs Cq pour fournir des données fiables qui représentent l'histoire biologique est présenté dans ce chapitre.
Dérivation de valeurs Cq précises<
Correction de la ligne de base
Une valeur Cq est déterminée pour chaque cible dans chaque échantillon. Différents progiciels d'analyse associés à différents instruments ont des approches différentes pour déterminer la valeur Cq ; (et utilisent également d'autres noms, par exemple Ct, Cp, point de décollage). Il n'entre pas dans le cadre de ce guide d'entrer dans les moindres détails de tous ces algorithmes. Cependant, les mesures qPCR basées sur les courbes d'amplification sont sensibles à la fluorescence de fond. La fluorescence de fond peut être causée par une série de facteurs, notamment le choix du matériel plastique, la fluorescence résiduelle de la sonde qui n'est pas éteinte, la fuite de lumière dans le puits d'échantillon et les différences dans la détection optique pour un puits de plaque de microtitration donné. Dans les essais bien conçus, le bruit de fond est faible par rapport au signal amplifié. Toutefois, la variation du signal de fond peut entraver la comparaison quantitative de différents échantillons. Il est donc important de corriger les variations de la fluorescence de fond qui entraînent des différences dans la ligne de base (Figure 10.1).
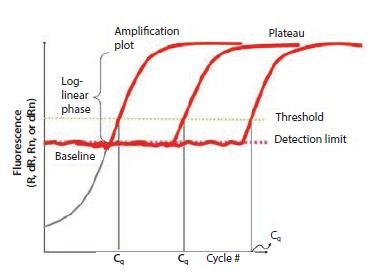
Figure 10.1Les composantes des courbes d'amplification. Ce graphique montre l'augmentation de la fluorescence avec le nombre de cycles pour différents échantillons. Le seuil est fixé au-dessus de la limite de détection mais bien en dessous de la phase de plateau pendant laquelle le taux d'amplification ralentit.
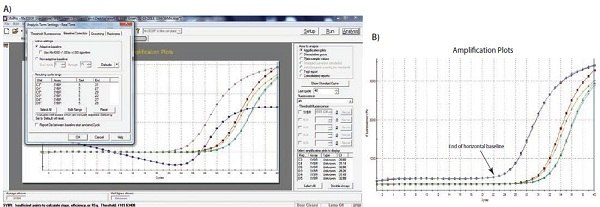
Figure 10.2A-B. A)Exemple typique de données tombant en dessous de la lecture de fluorescence normalisée zéro lorsque le réglage de la ligne de base est incorrect (tracé d'amplification bleu). B) Données brutes des mêmes tracés d'amplification montrant la limite de la ligne de base linéaire et que les données ne sont pas en cause.
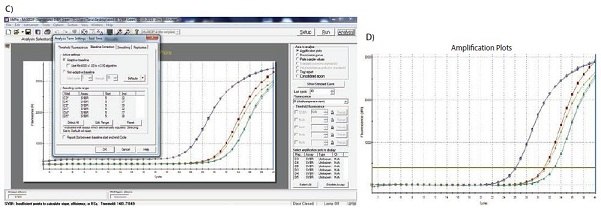
Figure 10.2C-D. C)Les limites du début et de la fin de la ligne de base sont définies à l'aide des paramètres appropriés du logiciel. D) L'application de la ligne de base corrigée permet d'obtenir des données de bonne qualité.
Setting the Threshold
Bien que certains chercheurs préconisent la cartographie des parcelles d'amplification individuelles pour estimer l'efficacité de l'amplification et les quantités cibles dans les échantillons mesurés2,3,4, l'approche originale et la plus courante pour calculer la Cq est d'utiliser un seuil. Le principe de la méthode du seuil est le suivant : pour visualiser le signal fluorescent associé à l'amplification qPCR, le signal doit augmenter jusqu'à dépasser la limite de détection de l'instrument (et donc la ligne de base ; figure 10.1). Le nombre de cycles nécessaires pour que cela se produise est proportionnel au nombre initial de copies de la cible dans l'échantillon. Par conséquent, plus de cycles sont nécessaires pour que le signal augmente au-dessus de la ligne de base si le nombre de copies initial est faible et moins de cycles si le nombre de copies est élevé. Étant donné que la ligne de base est fixée à la limite de détection du système, les mesures à la ligne de base seraient très imprécises. Par conséquent, plutôt que de mesurer l'intensité de la fluorescence minimale que le système peut détecter, une fluorescence plus élevée est sélectionnée et un seuil artificiel est introduit.
La sélection de l'intensité du seuil nécessite le respect de certains principes fondamentaux. Il est important que le seuil soit fixé à une intensité fixe pour une cible donnée et pour tous les échantillons à comparer. Si le nombre d'échantillons est trop important pour être placé sur une seule plaque, il convient d'adopter un schéma d'étalonnage interplaques, par exemple en incluant un contrôle répliqué qui sert de contrôle interplaques ou une dilution en série de la courbe standard. En théorie, le seuil peut être fixé n'importe où sur la phase log-linéaire de la courbe d'amplification. Toutefois, dans la pratique, la phase log-linéaire de l'amplification peut être perturbée par la dérive de la ligne de base de la fluorescence de fond, la phase de plateau ou les différences d'efficacité du test et, par conséquent, le gradient de la courbe d'amplification à des cycles plus élevés. Il est recommandé de fixer le seuil comme suit:
- Suffisamment au-dessus de la ligne de base de la fluorescence de fond pour éviter que le diagramme d'amplification ne franchisse le seuil prématurément en raison de la fluorescence de fond.
- Dans la phase logarithmique du tracé d'amplification où il n'est pas affecté par la phase de plateau (ceci est plus facilement visible en visualisant les tracés d'amplification sur une vue logarithmique, Figure 10.3A).
- A une position où les phases logarithmiques de tous les diagrammes d'amplification sont parallèles.
Le processus de fixation du seuil est illustré à la figure 10.3. Dans la figure 10.3A, les diagrammes d'amplification sont visualisés sur une échelle logarithmique de l'axe Y, ce qui permet d'obtenir une expansion visuelle de la phase logarithmique de l'amplification et de la présenter comme une partie linéaire du diagramme d'amplification. Le seuil est fixé à l'intensité de fluorescence la plus élevée (voir l'axe Y) qui se situe dans cette phase logarithmique et où tous les diagrammes d'amplification sont parallèles. L'échelle est ensuite ramenée à la vue linéaire (Figure 10.3B) montrant le réglage le plus élevé qui répond aux exigences de réglage du seuil. Le seuil peut également être fixé à l'extrémité inférieure de cette phase logarithmique (Figures 10.3C et 10.3D). Tant que la phase logarithmique des tracés d'amplification est parallèle, le ΔCq entre les échantillons n'est pas affecté par le réglage du seuil.
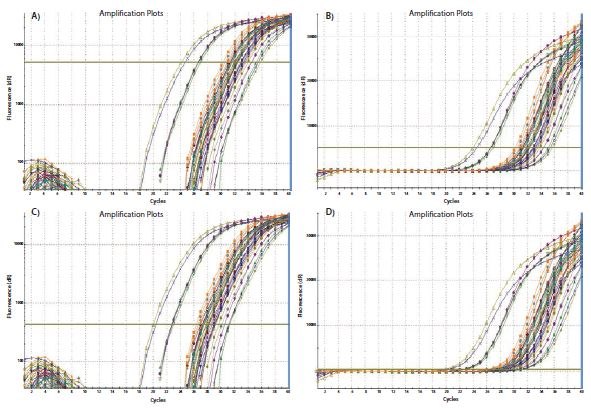
Figure 10.3Le réglage du seuil influe sur le Cq absolu enregistré et peut influencer le ΔCq entre les échantillons. A). En utilisant un tracé logarithmique vs linéaire des données, le seuil est fixé à l'intensité de fluorescence la plus élevée, mais où les tracés d'amplification montrent des phases logarithmiques parallèles. B). Le réglage du seuil est maintenu à partir de A) et est affiché sur le graphique linéaire vs linéaire. C). En utilisant un tracé logarithmique vs linéaire des données, le seuil est fixé à l'intensité de fluorescence la plus faible, mais où les tracés d'amplification montrent des phases logarithmiques parallèles. D). Le réglage du seuil est maintenu à partir de C) et est affiché sur le graphique linéaire vs linéaire. Dans chaque cas, les valeurs ΔCq entre les échantillons sont les mêmes.

Figure 10.4.L'analyse effectuée et démontrée dans la figure 10.3 a été répétée en utilisant un ensemble de données différent. Dans ce cas, les courbes d'amplification ne sont pas parallèles en raison d'une différence d'efficacité de la réaction à un Cq élevé. Les réglages les plus bas pour A) et B) donnent des valeurs ΔCq différentes de celles des réglages les plus élevés pour C) et D) (résumé dans le tableau 10.1).
Stratégies de quantification par qPCR
Un réglage précis de la ligne de base et du seuil est impératif pour une quantification fiable. Après avoir défini chacun de ces paramètres, une valeur Cq est générée et sert de base à la quantification. La quantité de cible dans un échantillon donné est alors déterminée à l'aide d'une courbe standard ou d'une quantification relative/comparative.
Quantification par courbe standard
Comme son nom l'indique, la quantification par courbe standard nécessite l'utilisation d'une courbe standard pour déterminer les quantités de cibles dans les échantillons de test. Toutes les quantités déterminées pour les échantillons sont donc relatives à la quantité attribuée à la courbe standard. Pour ce faire, il est nécessaire d'utiliser des étalons externes supplémentaires pour chaque série de réactions d'échantillons. Le choix du matériel pour la courbe standard est important pour éliminer les différences potentielles de quantification dues aux différences entre les efficacités de dosage dans les échantillons et dans les standards. Les sites de liaison des amorces des étalons externes doivent être les mêmes que ceux de la cible, contenir des séquences identiques à celles de la cible, avoir une complexité similaire et être manipulés d'une manière aussi similaire que possible. Par conséquent, lors de la mesure de la concentration d'une cible dans l'ADNc, il est préférable de mesurer le même ADNc dans une dilution en série d'un échantillon de contrôle. Toutefois, pour certaines études, des raisons pratiques s'y opposent ; il est donc important de reproduire les conditions de l'échantillon aussi fidèlement que possible, par exemple en ajoutant de l'ADNg d'une espèce non apparentée à l'espèce testée, à un oligonucléotide artificiel standard ou à un plasmide linéarisé portant la séquence standard. Une fois qu'une construction ou un amplicon approprié a été identifié, une courbe standard de dilutions en série est générée. La Cq pour la cible est déterminée pour chacun des étalons et tracée en fonction de la concentration ou de la concentration relative/du facteur de dilution sur une échelle logarithmique. Il en résulte une courbe standard qui est ensuite utilisée pour déterminer les concentrations des échantillons à tester en comparant les valeurs Cq dérivées de l'amplification des échantillons inconnus. Lors de l'utilisation d'une courbe standard pour la quantification, le réglage du seuil doit être maintenu constant pour la détermination de la Cq pour la norme et pour les échantillons sur la même plaque. Le seuil peut varier d'une plaque à l'autre.
Quantification relative/comparative
La quantification relative ou comparative utilise la différence de Cq comme déterminant des différences de concentration de la séquence cible dans différents échantillons. Plutôt que de mesurer les quantités de la cible par échantillon comme avec la méthode de la courbe standard, cela conduit à des ensembles de données montrant les changements de pli entre les échantillons.
Dans la forme originale de cette approche5, l'efficacité de tous les essais était supposée être de 100 %, ce qui permettait de supposer qu'un Cq différence de 1 (ΔCq = 1) était le résultat d'une différence de 2 fois dans la cible. Pour déterminer un changement de pli dans la cible ou le gène d'intérêt (GOI), les données doivent également être référencées par rapport à un contrôle de charge (gène de référence, ref ; voir ce qui suit pour une discussion concernant la normalisation des données).
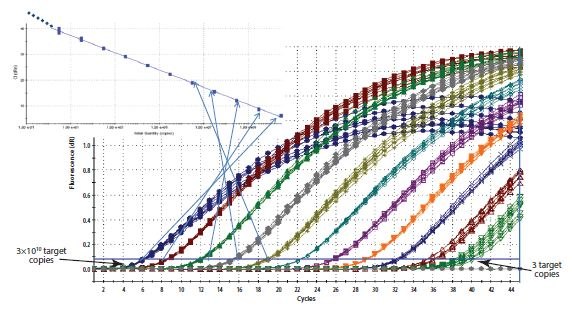
Figure 10.5.Construction d'une courbe standard. Le Cq enregistré pour chaque échantillon d'une série de dilutions est tracé sur une échelle logarithmique linéaire en fonction de la concentration relative.
.
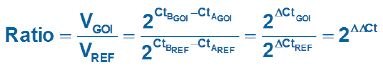
Équation 1.Original (Livak) Modèle de quantification relative.

Équation 2.Modèle de quantification relative adapté à l'efficacité (Pfaffl)
A titre d'exemple d'utilisation du modèle de quantification relative adapté à l'efficacité (équation 2), un ensemble de valeurs Cq est présenté dans le Tableau 10.2. L'efficacité pour le GOI est de 1,8 et pour le gène ref de 1,94.
L'efficacité pour le GOI est de 1,8 et pour le gène ref de 1,94.
Normalisation
Le principal objectif de la plupart des expériences basées sur la PCR est de répondre à la question fondamentale de savoir si la cible est présente dans l'échantillon (inconnue, UNK). Au niveau le plus simple, on répond à cette question en réalisant un gel et en examinant les fragments pour vérifier la présence ou l'absence du GOI souhaité. Lorsque le fragment est présent, la confirmation de la taille du fragment donne l'assurance d'un résultat positif. En revanche, en cas d'absence, il existe un risque de résultat faussement négatif. Il est donc essentiel de répéter l'essai et d'effectuer au moins une PCR supplémentaire pour servir de contrôle de chargement et de PCR positive. L'essai de contrôle d'inhibition universel, SPUD (voir Purification d'échantillon et évaluation de la qualité), peut être utilisé pour renforcer la confiance dans un résultat négatif. Une autre approche consiste à effectuer un test spécifique à un ou plusieurs gènes de référence. Traditionnellement, les tests PCR détectant les gènes de référence, la GAPDH, l'ARN ribosomique 18S ou la β-actine, étaient effectués en même temps que ceux du GOI et les fragments résultants étaient visualisés sur un gel. La GAPDH, l'ARN ribosomique 18S et la β actine sont exprimés de manière constitutive et ont donc été utilisés comme témoins de charge dans les analyses semi-quantitatives. Cependant, il est rapidement apparu que ces gènes ne sont pas exprimés de manière ubiquitaire à la même concentration dans toutes les cellules, quel que soit le protocole expérimental. Par conséquent, le besoin d'une référence stable s'est fait sentir lorsque l'objectif était de mesurer les concentrations relatives d'acides nucléiques, généralement l'ADNc, mais aussi l'ADNg lorsque, par exemple, on examine la variation du nombre de copies d'un gène. Il existe de nombreuses méthodes de normalisation des différences techniques, ce qui signifie que l'approche appropriée pour l'expérience spécifique doit être sélectionnée et validée7. Il est essentiel de reconnaître que l'adoption de techniques de normalisation inappropriées peut être plus préjudiciable au processus analytique global que l'absence de normalisation8.
L'effet de la qualité de l'échantillon sur la normalisation de l'essai
L'effet de l'intégrité et de la pureté de l'échantillon sur les mesures de la quantité cible par qPCR et RT-qPCR a été longuement discuté (Purification de l'échantillon et évaluation de la qualité, Contrôle de la qualité de l'échantillon et Transcription inverse, Transcription inverse). Il a été démontré que les inhibiteurs présents dans l'échantillon et la dégradation de l'ARN ont un effet différentiel sur la mesure d'une cible donnée9. Les inhibiteurs affectent la mesure de toute cible, mais à un degré différent, en fonction de la conception de l'essai. La dégradation de l'ARN total affecte la mesure de l'ARNm et de l'ARNm10, ce qui dépend encore une fois fortement de la conception expérimentale globale. Il est donc essentiel de tenir compte de l'effet de la concentration de la matrice sur la réaction de RT et de l'effet de la qualité de l'échantillon sur les données après normalisation. La normalisation ne contrera pas l'effet des essais ou des échantillons de faible qualité (voir Optimisation et validation de l'essai).
Approches de normalisation
En principe, les méthodes de normalisation neutralisent la variabilité qui peut être introduite au cours du processus en plusieurs étapes nécessaire à la réalisation d'une analyse qPCR (Figure 10.6). Cependant, l'application de la normalisation à une étape quelconque du processus peut ne pas contrôler l'erreur technique et/ou le biais qui a été, ou sera, introduit à une étape antérieure ou ultérieure, respectivement. Les méthodes de normalisation ne s'excluent pas mutuellement et il est donc recommandé d'adopter une combinaison de contrôles11.
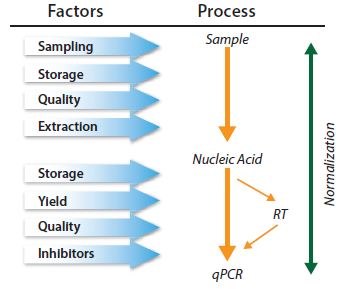
Figure 10.6.La qPCR est un processus à plusieurs étapes et chaque étape doit être contrôlée. La normalisation doit être envisagée dans le cadre d'une série de contrôles.
Sélection des gènes de référence
Les gènes de référence sont des cibles dont la quantité ne change pas à la suite de l'expérience. Lors de la quantification de la variation du nombre de copies d'ADN, dans laquelle le nombre de copies de la séquence d'intérêt peut changer, la mesure est simplement normalisée en ciblant une région génomique alternative dont on sait qu'elle ne changera pas. Un exemple d'application de ce principe est la mesure de l'amplification génomique du récepteur 2 du facteur de croissance épidermique humain (HER-2)13. L'instabilité génomique HER-2 est un indicateur pronostique dans le cancer du sein et la mesure précise du statut d'amplification HER-2 est importante pour la prise en charge des patientes. Le statut HER-2 peut être mesuré par qPCR en comparant les copies de HER-2 avec une autre cible génomique qui sert de contrôle.
Lorsque l'on mesure l'expression génique, les gènes de référence sont des cibles dont les concentrations d'ARNm ne changent pas à la suite de l'expérience. Un exemple d'étude serait une étude dans laquelle l'effet sur l'expression du gène X est mesuré après l'ajout d'un composé mitogène à une monocouche cellulaire. Un point de référence est nécessaire pour mesurer le changement dans le gène X. Par conséquent, un autre gène (ou d'autres gènes) dont on sait qu'il n'est pas affecté par le mitogène en question est également mesuré. Le chercheur est ainsi confronté au défi immédiat de trouver une cible d'ARNm qui n'est pas affectée par la procédure expérimentale, avant de pouvoir étudier le RGO. Ce processus de validation des gènes de référence est fondamental pour une mesure précise du RGO. L'approche la plus répandue de la normalisation consiste à ignorer ce processus et à normaliser les données d'expression génique par rapport à un seul gène de référence non validé. Cette pratique n'est pas recommandée et est en opposition directe avec les lignes directrices MIQE1. La quantification de l'ARNm par RT-qPCR a été régulièrement compromise par le choix incorrect des gènes de référence. Il n'est pas acceptable de suivre les pratiques relativement courantes consistant à utiliser un gène de référence parce que les amorces se trouvent déjà dans le congélateur, qu'il a été utilisé par le passé sur des Northern blots, qu'il est utilisé par un collègue ou qu'il a été utilisé dans un autre laboratoire pour une expérience différente. Les gènes de référence doivent être validés dans le cadre de scénarios expérimentaux spécifiques afin de s'assurer que le gène de référence en question n'est pas affecté par l'expérience. Si cette validation n'est pas effectuée et que le gène de référence est affecté par l'expérience, les résultats peuvent être incorrects et les interprétations ultérieures risquent d'aboutir à des données dépourvues de sens8.
Il existe toute une série de publications scientifiques décrivant différentes méthodes de normalisation7-14 ainsi qu'une pléthore de publications décrivant les protocoles requis pour identifier les gènes normalisateurs les plus appropriés pour un scénario expérimental donné. La sélection de gènes de référence stables exige que l'analyste évalue la stabilité de la qPCR pour un certain nombre (généralement 10 à 20 gènes) de cibles d'ARNm candidats7 sur un sous-ensemble d'échantillons qui représentent les ARNm de test et de contrôle. Un protocole complet est fourni dans Annexe A, Protocoles, de ce guide et peut être utilisée en combinaison avec différentes méthodes analytiques utilisant des programmes tels que REST15, GeNorm14, Bestkeeper16, ou NormFinder17. Cette procédure est décrite plus en détail dans la section suivante, Analyse de la stabilité du gène de référence.
Analyse de la stabilité du gène de référence
Le gène de référence est littéralement le point pivot des essais de quantification relative par qPCR. Il est donc essentiel pour la fiabilité de l'ensemble de l'essai que le gène de référence soit stable. Si l'expression du gène de référence varie d'un échantillon à l'autre, la variation sera directement transférée aux résultats de la quantification et la variabilité ajoutée peut masquer l'effet biologique observable souhaité ou, pire encore, créer une apparence entièrement artificielle d'effet biologique, qui n'est pas lié au gène d'intérêt réel. Pour ces raisons, il est fortement recommandé de suivre plusieurs mesures de sécurité afin de rendre la variabilité des gènes de référence insignifiante et de rendre les mesures des effets biologiques aussi significatives que possible.
La mesure de sécurité la plus importante est sans doute l'utilisation non pas d'un seul, mais de deux gènes de référence ou plus. L'expression de plusieurs gènes de référence peut être moyennée pour réduire la variabilité technique due à la normalisation. Cela peut être utile pour améliorer la signification des mesures de petits effets biologiques. Cependant, plus important encore, deux gènes de référence ou plus fournissent des contrôles mutuels pour maintenir la stabilité et contrôler les événements inattendus qui peuvent influencer les niveaux d'expression de l'un des gènes de référence. Avec un seul gène de référence, il existe un risque que des influences inattendues de l'expression génique ne soient pas détectées dans l'essai.
Une autre mesure de sécurité consiste à utiliser plusieurs méthodes pour identifier les gènes de référence stables. L'exemple suivant illustre plusieurs aspects de la normalisation des gènes de référence, y compris un avantage possible de l'utilisation des méthodes geNorm et NormFinder sur le même ensemble de données.
Tableau 10.3 contient une liste de gènes de référence candidats qui ont été évalués au cours d'un atelier que nous avons précédemment organisé avec l'EMBL. Les échantillons ont été collectés à partir d'une culture de cellules humaines dans deux groupes de traitement différents. Cet ensemble de données sera utilisé pour démontrer certains aspects de la validation des gènes de référence
Les algorithmes NormFinder et geNorm ont tous deux été mis au point en partant de l'hypothèse que l'essai d'une multitude de gènes de référence candidats peut être utilisé pour classer la stabilité des gènes de référence candidats individuels. Cette hypothèse peut se vérifier si, par exemple, tous les gènes de référence candidats varient de manière stochastique autour de niveaux d'expression stables. Toutefois, ce n'est pas nécessairement le cas dans la réalité. Pour éviter des résultats trompeurs, il est donc prudent d'éviter les candidats gènes de référence régulés et, en particulier, corégulés.
La liste des gènes de référence candidats figurant dans le tableau 10.3 a été spécifiquement choisie pour sélectionner des gènes appartenant à des classes fonctionnelles différentes, réduisant ainsi le risque de corégulation des gènes. Une exception notable est la GAPDH, qui est présente ici en deux versions. Bien que cela n'affecte pas cette analyse, la meilleure pratique consiste à éviter les entrées multiples de gènes susceptibles d'être corégulés.
Le premier algorithme présenté est geNorm. Il fournit une évaluation de la stabilité des gènes en calculant une mesure de stabilité des gènes appelée valeur M, qui est basée sur des comparaisons par paire entre le candidat gène de référence analysé et tous les autres candidats gènes de référence dans l'ensemble de données. Elle est effectuée de manière itérative, ce qui signifie que dans cet exemple, la procédure est d'abord effectuée sur les 15 gènes de référence candidats, le moins stable est éliminé, le processus est répété sur les 14 restants, le deuxième candidat le moins stable est éliminé, et ainsi de suite jusqu'à ce qu'il reste deux gènes de référence.
Il peut y avoir des moments où l'identification du gène de référence le plus stable peut être particulièrement difficile. C'est le cas lorsque tous les gènes de référence candidats sont peu performants. Un autre cas peut être celui où tous les gènes de référence candidats sont performants. Pour faire la distinction entre ces deux cas, il est utile de préciser que les gènes de référence dont la valeur M est inférieure à 0,5 peuvent être considérés comme exprimés de manière stable.
Le deuxième algorithme présenté est NormFinder, un progiciel d'analyse des gènes de référence disponible gratuitement (annexe B, ressources supplémentaires). L'algorithme sous-jacent adopte une approche de type ANOVA pour l'évaluation de la stabilité des gènes de référence, en ce sens que l'ensemble et les sous-groupes sont analysés pour déterminer les variations. L'un des avantages de cette méthode est que les mesures obtenues sont directement liées aux niveaux d'expression des gènes. Un écart-type de 0,20 en Cq unités représente donc environ 15 % de variation dans les niveaux d'expression du nombre de copies du gène de référence candidat particulier.
Pour des raisons de commodité, dans cette démonstration, ces deux progiciels d'analyse sont accessibles à l'aide du logiciel d'analyse de données GenEx (MultiD), mais ils sont également disponibles en tant que progiciels indépendants (annexe B, ressources supplémentaires).
Les diagrammes à barres présentés dans la figure 10.7 illustrent les gènes de référence classés en fonction de leurs mesures de stabilité respectives à l'aide des deux algorithmes. En outre, un graphique montrant l'écart-type cumulé de NormFinder indique qu'une combinaison des trois meilleurs gènes de référence peut améliorer la stabilité.

Figure 10.7.Diagrammes à barres montrant les mesures de stabilité : Valeurs M pour geNorm et écarts types pour NormFinder. En outre, un graphique montrant l'écart-type cumulé de NormFinder indique qu'une combinaison des trois meilleurs gènes de référence peut améliorer la stabilité. L'ensemble de données a été généré à partir d'essais conçus pour les gènes de référence candidats présentés dans le tableau 10.3 et mesurés sur une culture de cellules humaines dans deux groupes de traitement différents. Remarquez que, dans ce cas, les algorithmes de stabilité des gènes de référence geNorm et NormFinder ne sont pas d'accord sur les meilleurs gènes de référence.
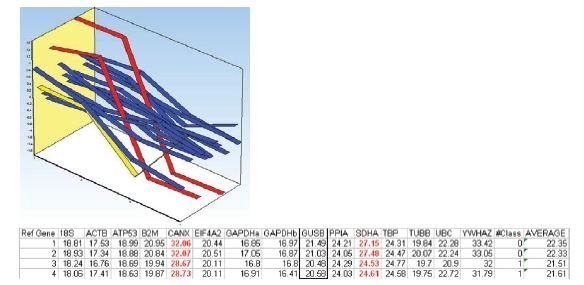
Figure 10.8.Profil d'expression centré moyen des gènes candidats de référence des deux échantillons dans chaque groupe de traitement. Les échantillons 1 et 2 appartiennent au premier groupe de traitement et les échantillons 3 et 4 appartiennent au deuxième groupe de traitement. Les profils d'expression de SDHA et CANX sont indiqués en rouge. Le profil d'expression de l'UBC est indiqué en jaune. Le tableau énumère les valeurs Cq mesurées dans l'ensemble des données.
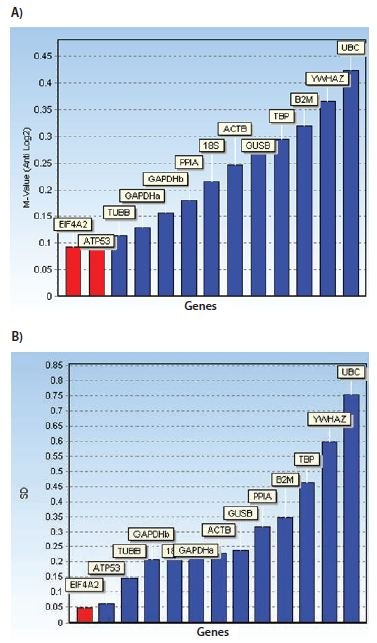
Figure 10.9.L'inspection des profils d'expression et des valeurs Cq mesurées (figure 10.8) a fait craindre que SDHA et CANX ne soient corégulés dans l'essai appliqué. Cette corégulation peut perturber les algorithmes de stabilité des gènes de référence. Diagrammes à barres montrant les mesures de stabilité : A) valeurs M pour geNorm et B) écarts types pour NormFinder. L'ensemble de données est le même que celui utilisé dans la figure 10.8, à l'exception des données pour SDHA et CANX qui ont été supprimées. Remarquez qu'avec cet ensemble de données réduit, les algorithmes de stabilité des gènes de référence geNorm et NormFinder s'accordent sur les meilleurs gènes de référence.
Méthodes de normalisation alternatives
La normalisation par rapport aux gènes de référence est la méthode la plus courante.Bien que la normalisation par rapport aux gènes de référence soit la méthode la plus courante pour la normalisation des essais, il existe des situations où cette approche n'est pas appropriée, par exemple lorsqu'il s'agit de comparer un grand nombre de gènes dans un groupe hétérogène d'échantillons, ou lors du profilage des ARNm. Dans ces scénarios, il est nécessaire d'adopter une autre stratégie.
Normalisation en fonction de la masse tissulaire ou du nombre de cellules
La mesure du nombre de cellules ou de la masse tissulaire à utiliser comme facteur de normalisation n'est pas aussi simple qu'il n'y paraît à première vue. Les expériences de culture cellulaire sont relativement faciles à normaliser sur la base du nombre de cellules. Cependant, l'ajout d'un traitement peut avoir un impact sur la morphologie des cellules, ce qui complique le rapport entre le nombre de cellules et l'ARN total/les gènes exprimés par rapport à une culture témoin. Le traitement expérimental peut entraîner la production d'une matrice cellulaire supplémentaire, ce qui entraîne des différences dans l'efficacité de l'extraction des acides nucléiques.
Les tissus biologiques peuvent être très hétérogènes au sein d'un même sujet et d'un sujet à l'autre, une plus grande variation étant apparente lorsque des tissus sains sont comparés à des tissus malades. Même des tissus apparemment moins complexes, tels que le sang, peuvent différer considérablement en termes de nombre de cellules et de composition, de sorte que l'expression des gènes varie considérablement entre des donneurs apparemment sains18.
Tout retard dans les processus utilisés pour purifier l'acide nucléique entraînera des altérations dans l'ARN mesuré. Par exemple, les retards dans le traitement des cellules mononucléaires du sang périphérique et dans l'extraction de l'ARN des cellules entraînent des changements considérables dans l'expression des gènes19. Les méthodes sous-jacentes aux procédures d'extraction sont également des sources majeures de variation technique. Même le processus d'isolement choisi pour l'échantillonnage des cellules dérivées du sang et la purification de l'ARN entraînent des différences dans les profils d'expression génique apparents20. Par conséquent, la première considération en matière de normalisation est de s'assurer que la collecte et le traitement sont absolument identiques pour tous les échantillons. Il est ensuite essentiel d'effectuer un contrôle de qualité suffisant pour être certain de la concentration, de l'intégrité et de la pureté de l'échantillon (Purification de l'échantillon et évaluation de la qualité et les protocoles associés dans Annexe A).
Normalization to RNA Concentration
As a minimum, an estimation of template concentration (DNA for qPCR or RNA for RT-qPCR) is important and, as mentioned in Purification d'échantillons et évaluation de la qualité, il est essentiel de s'assurer que le même instrument est utilisé pour toutes les mesures car la détermination de la concentration d'acide nucléique est également variable et dépendante de la technique.
Lorsque l'on mesure la concentration d'ARN total, la grande majorité de l'échantillon est composée d'ARNr, seule une petite fraction étant constituée de l'ARNm d'intérêt lors de l'examen de l'expression génique, ou de l'ARNnc lors de l'examen de la régulation de l'expression génique. Cela signifie que si la concentration d'ARNr augmente légèrement mais que l'ARNm reste constant, la concentration d'ARN total augmentera. La concentration d'ARNm doit augmenter de manière significative pour provoquer une augmentation apparente de la concentration d'ARN total. Par conséquent, la concentration d'ARNr n'est pas une mesure fiable de la concentration d'ARNm, mais pour de nombreux protocoles, une concentration égale d'ARN est nécessaire pour garantir une transcription inverse précise (voir Transcription inverse).
Normalisation à l'expression génique globale
Lorsque l'on mesure un grand nombre de cibles, l'analyste peut estimer la moyenne globale de l'expression génique totale et identifier les séquences d'ARN régulées qui s'écartent de cette moyenne. Cette approche est classiquement utilisée pour la normalisation des réseaux d'expression génique. Elle constitue une alternative intéressante à l'utilisation de gènes de référence et peut être préférable lorsque de nombreuses cibles sont mesurées.
Une autre approche récemment explorée consiste à mesurer les éléments répétitifs exprimés de manière endogène (ERE) qui sont présents dans de nombreux ARNm. De nombreuses espèces contiennent ces éléments répétés (ALU chez les primates, éléments B chez les souris), qui peuvent fournir une estimation de la fraction d'ARNm. Il a été démontré que la mesure de ces séquences cibles fonctionne comme les systèmes de normalisation conventionnels9 ;(Le Bert, et al., en préparation) et peut offrir une solution universelle ou une alternative pour les expériences complexes où des combinaisons de gènes de référence stables ne sont pas disponibles.
Normalisation des données miARN
A ce jour, il n'y a pas eu de rapports sur un gène de référence universel pour les miARN. Par conséquent, la sélection du système de normalisation est encore assez empirique. Lorsque cela est possible, des miARN invariants stables peuvent être identifiés à partir d'approches à l'échelle du génome, c'est-à-dire des puces à ADN. Les petits ARN nucléolaires (snoRNA) ont également été utilisés comme gènes de référence. L'expression globale des gènes est également une méthode utile pour normaliser l'expression des miARN lorsqu'une référence stable est inconnue et que plusieurs centaines de cibles ont été analysées21,22,23. Cette méthode est plus appropriée pour ceux qui utilisent des approches permettant de capturer tous les miARN sous forme d'ADNc dans une forme multiplexée, par ex, Exiqon et miQPCR (voir Castoldi et al. dans PCR Technologies, Current Innovations24).
Réplicats biologiques et techniques
L'objectif de la normalisation est d'éviter les erreurs systématiques et de réduire la variabilité des données en vue d'une éventuelle analyse statistique. Un autre aspect important de la préparation des données pour l'analyse statistique est l'utilisation de réplicats de données.
Les réplicats biologiques sont absolument nécessaires pour l'analyse statistique. Les niveaux de signification statistique sont souvent fixés à un seuil de signification de 5 %. Pour les effets biologiques proches d'un tel niveau de signification, il peut être nécessaire d'avoir au moins 20 réplicats biologiques pour déterminer le niveau de signification des essais (1:20 correspondant à 5 %). En fait, il a été suggéré qu'au moins 50 fois le nombre d'observations doivent être enregistrées pour une estimation précise de la signification25, c'est-à-dire de l'ordre d'un millier d'échantillons biologiques. Naturellement, les limitations pratiques permettent rarement d'effectuer des répétitions biologiques à ces niveaux. En outre, les estimations précises du nombre de répétitions biologiques nécessaires pour atteindre un niveau de signification donné dépendent également du niveau de variabilité des données. Néanmoins, il est important de comprendre qu'une erreur courante consiste à sous-estimer le nombre de répétitions biologiques nécessaires pour parvenir à des conclusions fiables. Il est recommandé de réaliser une étude pilote initiale pour évaluer la variabilité inhérente à l'essai et la taille potentielle de l'effet biologique observable afin de disposer d'une bonne base pour estimer le nombre nécessaire de réplicats biologiques26.
Les réplicats techniques ne sont pas utilisés directement pour l'analyse statistique. Ils servent plutôt à sauvegarder les échantillons (au cas où certains échantillons seraient perdus au cours du processus de manipulation technique) et à améliorer l'évaluation de la précision des données. Les réplicats techniques peuvent améliorer la précision des données si l'on suppose qu'ils varient de manière stochastique autour de la mesure exacte à chaque étape du processus de traitement technique. La moyenne des répliques techniques est plus proche de la mesure exacte. L'effet du calcul de la moyenne des réplicats techniques peut être illustré en notant la taille de l'intervalle de confiance dans un ensemble de données simulées avec une variabilité prédéterminée, c'est-à-dire un écart type fixé à un. Comme le montre le tableau 10.4, l'intervalle de confiance se réduit avec l'augmentation du nombre de réplicats techniques (échantillons), ce qui indique une estimation plus précise de la mesure exacte. En outre, le rétrécissement de l'intervalle de confiance est le plus spectaculaire lorsque le nombre de réplicats techniques est faible. En augmentant le nombre de réplicats de 2 à 3, l'intervalle de confiance passe de 8,99 à 2,48, soit une amélioration de la précision de l'estimation de la mesure exacte d'un facteur de plus de 3. Si les répétitions supplémentaires continuent d'améliorer l'estimation de la précision de la mesure, l'effet est de moins en moins important. Par conséquent, il est évident que dans les cas où la variabilité de la manipulation technique est un problème, il peut être très avantageux d'utiliser des triplicats plutôt que des duplicats.
Les résultats de l'analyse de la variabilité de la manipulation technique sont présentés ci-dessous.
Analyse statistique et visualisation des données
L'analyse scientifique des données biologiques est centrée sur la formulation et la vérification d'hypothèses. La formulation d'une hypothèse nécessite une compréhension détaillée des conditions et des variables de l'essai. La vérification réussie d'une hypothèse implique une exécution minutieuse et une conception expérimentale appropriée pour maximiser le signal observable souhaité tout en minimisant la variabilité technique. Dans ce contexte, il est utile de faire la distinction entre les études exploratoires et les études de confirmation (Figure 10.10).
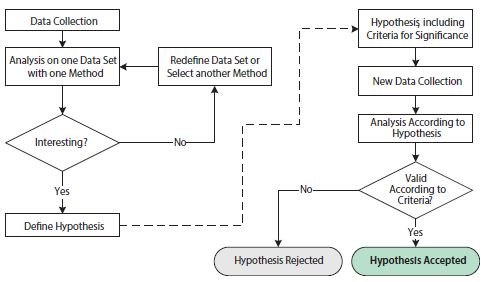
Figure 10.10.Organigramme illustrant les opérations impliquées dans les analyses statistiques exploratoires et confirmatoires. La partie gauche de la figure, avant la flèche en pointillés, montre les opérations d'une étude statistique exploratoire. La partie droite de la figure, après la flèche en pointillés, illustre les opérations d'une étude statistique confirmatoire.
Les exigences d'une étude de confirmation sont basées sur des critères statistiques beaucoup plus stricts. Tout d'abord, l'hypothèse de l'étude, y compris les critères de signification, doit être définie avant la collecte des données et avant l'analyse. En outre, l'ensemble des données à analyser doit avoir été collecté exclusivement à cette fin. Il est statistiquement incorrect de réutiliser l'ensemble des données de l'étude exploratoire dans l'étude confirmatoire, car cet ensemble de données favoriserait intrinsèquement l'hypothèse proposée. Le résultat final de l'étude confirmatoire est une hypothèse rejetée ou acceptée selon les critères préétablis.
Statistical Tests
Pour les tests statistiques, la probabilité qu'un phénomène observé se produise par hasard est analysée. C'est ce qu'on appelle l'hypothèse nulle28. Si le phénomène observé est rare selon l'hypothèse nulle, la conclusion est qu'il est peu probable que l'hypothèse nulle soit valide. L'hypothèse nulle est rejetée et la probabilité que l'hypothèse alternative soit significative est acceptée.
La probabilité estimée que le phénomène observé se soit produit par hasard est appelée la valeur p. La valeur p est mesurée dans une fourchette de 0 à 1, ou de manière équivalente, en unités de pourcentage. Les critères statistiques d'une étude confirmatoire comprennent un seuil alpha en dessous duquel les valeurs calculées indiquent que le phénomène observé est significatif. Un seuil alpha de 5 % est couramment utilisé, bien qu'il doive être ajusté pour correspondre aux critères souhaités et nécessaires qui sont spécifiques à l'objet de l'étude.
De nombreux algorithmes ont été développés pour calculer p les valeurs sous diverses hypothèses et à des fins différentes. Un algorithme courant est le test t de Student. Le test t de Student est utilisé pour calculer une valeur p basée sur la différence entre les valeurs moyennes de deux groupes de données. L'hypothèse principale du test t de Student est que les deux groupes de données sont indépendants et conformes à des distributions normales. Le test t de Student présente l'avantage d'être puissant par rapport aux tests statistiques non paramétriques29. Un test non paramétrique équivalent au test t de Student peut être l'un des tests statistiques non paramétriques les plus connus : le test des rangs de Wilcoxon (parfois appelé test U de Mann-Whitney ; à ne pas confondre avec le test des rangs signés de Wilcoxon qui est utilisé pour comparer deux groupes appariés). Les tests statistiques non paramétriques, tels que le test des rangs de Wilcoxon, présentent l'avantage par rapport aux tests statistiques paramétriques, tels que le test t de Student, de ne pas dépendre d'hypothèses préalables sur les distributions des ensembles de données. Un test de Kolmogorov-Smirnov pour la distribution normale peut être utilisé pour décider s'il faut appliquer le test t de Student ou l'un des tests non paramétriques
En plus du choix de l'algorithme pour& nbsp;p
l'analyse des données, le test de Wilcoxon et le test des rangs ont l'avantage d'être paramétriques.nbsp;p, les ensembles de données qui sont introduits dans l'algorithme de calcul de la valeur p peuvent être manipulés pour faciliter l'observation des propriétés souhaitées dans l'ensemble de données. La combinaison des étapes de manipulation des données brutes et du choix de l'algorithme de calcul de la valeur p fait partie de la construction d'un modèle d'hypothèse.
La construction de modèles d'hypothèse dans la phase exploratoire d'une analyse statistique offre un degré élevé de liberté et constitue une partie importante de la recherche scientifique. Cependant, une hypothèse n'est jamais prouvée à l'aide d'une approche scientifique et statistique. Une approche scientifique correcte consiste à formuler une hypothèse nulle, à utiliser un ensemble de données indépendantes (de préférence nouvellement collectées) et à accepter ou rejeter l'hypothèse nulle conformément à l'organigramme de l'étude confirmatoire (figure 10.10).
Techniques de visualisation pour l'analyse univariée
Tout comme il existe de nombreuses méthodes d'analyse, il existe également de nombreuses techniques de visualisation des données parmi lesquelles choisir. Pour l'analyse de données univariées, un simple diagramme à barres avec les barres d'erreur associées est une technique de visualisation appropriée. Bien qu'il s'agisse d'une technique de visualisation simple et courante, certains points méritent d'être soulignés. Tout d'abord, les barres d'erreur peuvent illustrer différentes sources de variabilité : la variabilité inhérente aux données (l'écart-type, ET) ou la précision avec laquelle la valeur moyenne a été déterminée. Deuxièmement, la précision avec laquelle la valeur moyenne a été déterminée peut être illustrée de différentes manières, mais elle dépend en fin de compte d'une combinaison de la variabilité inhérente des données avec le nombre d'échantillons (N) et dans sa forme brute, elle est appelée l'erreur standard de la moyenne (SEM, Equation 1):
Les barres d'erreur peuvent illustrer différentes sources de variabilité : la variabilité inhérente des données (l'écart-type) ou la précision avec laquelle la valeur moyenne a été déterminée.
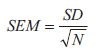
Équation 1.SEM
L'intervalle de confiance (IC, équation 2) est une mesure de la précision de la moyenne.
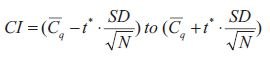
Équation 10-2.Cl
La figure 10.11 montre un graphique à barres avec des barres d'erreur indiquant l'intervalle de confiance à 95 % dans chaque groupe expérimental, mettant en évidence l'incertitude associée à l'estimation moyenne pour un exemple d'expression génique dans des échantillons provenant de différents organes après un traitement avec plusieurs doses de médicament. En outre, les valeurs de signification statistique du test t sont indiquées pour la différence d'expression génique entre les échantillons de contrôle et chacun des trois échantillons différents provenant de réponses à différentes doses de médicaments, indiquées au moyen d'un astérisque. Il est d'usage qu'un astérisque corresponde à une valeur inférieure à 0,05, deux astérisques correspondent à une valeur inférieure à 0,01 et trois astérisques correspondent à une valeur inférieure à 0,001.
Les valeurs de signification sont indiquées dans le tableau ci-dessous pour la différence d'expression génique entre les échantillons de contrôle et chacun des trois échantillons de réponses différentes à la dose de médicament.
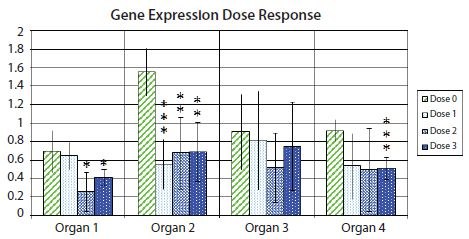
Figure 10.11.Changement de pli (log2) de l'expression d'un gène d'intérêt par rapport à une paire de gènes de référence, par rapport à l'expression dans l'échantillon ayant l'expression la plus faible dans chaque type d'organe. La hauteur des barres indique l'expression moyenne du gène dans plusieurs échantillons dans des groupes d'échantillons non traités (dose 0) ou d'échantillons traités à l'une des trois doses différentes de médicament (dose 1, dose 2 et dose 3). Les barres d'erreur indiquent les estimations de l'intervalle de confiance à 95 % des expressions moyennes. Un astérisque indique une différence statistiquement significative entre les moyennes d'un ensemble d'échantillons traités par rapport à la moyenne de l'ensemble d'échantillons non traités à 5 % ; deux astérisques indiquent une différence statistiquement significative à 1 % ; trois astérisques indiquent une différence statistiquement significative à 0,1 %.
La valeur de 0,05 pourrait être tout aussi significative qu'une valeur de 0,051.
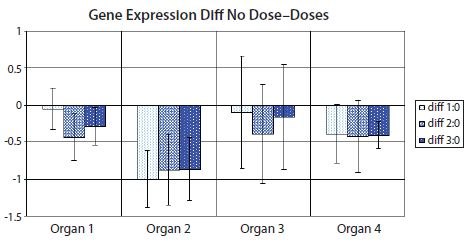
Figure 10.12.Diagramme à barres montrant la différence entre les moyennes de l'ensemble d'échantillons non traités (dose 0) et de l'un des ensembles d'échantillons traités (dose 1, dose 2 ou dose 3) dans l'ensemble de données de la figure 10.11. Les barres d'erreur indiquent l'intervalle de confiance de la différence entre les moyennes. Les barres d'erreur qui ne traversent pas l'axe des x indiquent que la comparaison des moyennes correspondantes est statistiquement significative à 5 % dans un test t. PCR Technology, Current Innovations-3rd ed. by Taylor and Francis Group LLC Books. Reproduit avec l'autorisation de Taylor and Francis Group LLC Books dans le format de réutilisation dans un livre/e-book via le Copyright Clearance Center.
Hierarchical Clustering
L'une des méthodes les plus simples et les plus utiles pour caractériser les données consiste à les représenter sous la forme d'un nuage de points (par exemple, en représentant la valeur mesurée de Cq mesurées d'un gène par rapport aux valeurs Cq correspondantes d'un autre gène pour un ensemble d'échantillons biologiques dans un diagramme 2D). Les tracés en une ou deux dimensions sont facilement visualisables par l'œil humain. Les tracés en trois dimensions sont également possibles avec des outils appropriés, mais les tracés à plus haute dimension sont beaucoup plus difficiles à visualiser. Toutefois, pour les études exploratoires, l'ensemble de données est intrinsèquement multidimensionnel et les diagrammes de dispersion de l'ensemble des données peuvent donc s'avérer peu pratiques. Dans un ensemble de données qPCR, il peut y avoir, par exemple, plusieurs gènes et/ou plusieurs types d'échantillons biologiques représentés.
Une autre façon populaire de caractériser et de visualiser les données des études exploratoires consiste à analyser les mesures de distance entre les points de données dans le nuage de points. Il existe différentes mesures de distance, notamment les corrélations euclidiennes, de Manhattan et de Pearson. Grâce à la puissance de calcul, il est facile de calculer les distances, même pour des données multidimensionnelles dont la dimensionnalité est bien supérieure à trois dimensions. Pour le regroupement hiérarchique agglomératif, le processus itératif suivant est exécuté : 1) Trouver les deux objets les plus proches et les fusionner en un cluster ; 2) Définir le nouveau cluster comme un nouvel objet par le biais d'une méthode de clustering ; 3) Répéter à partir de 1) jusqu'à ce que tous les objets aient été combinés en clusters30. Parmi les méthodes de regroupement, on peut citer la méthode de Ward, le lien unique et le lien moyen31. Un dendrogramme est souvent utilisé pour visualiser les résultats du regroupement hiérarchique.
L'interprétation des dendrogrammes de regroupement hiérarchique des données qPCR permet souvent de tirer des conclusions sur les similitudes entre les profils d'expression génique. Dans une étude exploratoire, ces similitudes peuvent alors être utilisées pour formuler des hypothèses sur la corégulation de l'expression génique, qui peuvent être acceptées ou rejetées dans des études confirmatoires ultérieures. Les avantages des dendrogrammes de regroupement hiérarchique sont notamment la clarté avec laquelle les relations de similarité sont visualisées. D'un autre côté, l'accent mis sur les mesures de similarité peut être perçu comme limitant la formulation d'hypothèses, car des profils d'expression similaires peuvent être des attributs redondants dans les hypothèses. Il peut être plus intéressant d'identifier des ensembles de profils d'expression qui se complètent dans une combinaison spécifique, pour répondre à l'hypothèse souhaitée.
Analyse en composantes principales
Il s'agit d'une autre méthode populaire pour caractériser les profils d'expression.Une autre façon courante de caractériser et de visualiser les données provenant d'études exploratoires consiste à tirer parti des informations contenues dans l'ensemble des données multidimensionnelles, à sélectionner les propriétés souhaitées et à les projeter sur un nuage de points de dimension inférieure, tel qu'un graphique en 2D ou en 3D. Pour ce faire, on peut utiliser l'analyse en composantes principales (ACP)32,33,34, 35. Ici, le système de coordonnées original de l'ensemble de données (c'est-à-dire les profils d'expression mesurés par qPCR) est transformé en un nouvel espace multidimensionnel où de nouvelles variables (composantes principales : PC ou facteurs) sont construites. Chaque PC est une combinaison linéaire des sujets de l'ensemble de données original. Par définition mathématique, les PC sont extraites par ordre successif d'importance. Cela signifie que la première PC explique la majeure partie de l'information (variance) présente dans les données, la deuxième moins et ainsi de suite. Par conséquent, les deux ou trois premières coordonnées des PC (appelées scores) peuvent être utilisées pour obtenir une projection de l'ensemble des données sur une dimension commodément réduite, adaptée à la visualisation sur un graphique en 2D ou en 3D. En utilisant les deux ou trois premiers PC pour la représentation, on obtient la projection qui tient compte de la plus grande variabilité de l'ensemble de données. La variance due aux conditions de la conception expérimentale devrait être systématique, tandis que la variance de confusion devrait être aléatoire, de sorte que cette représentation peut être souhaitée dans des conditions appropriées.
Comme indiqué précédemment pour le regroupement hiérarchique, l'interprétation de l'ACP de la qPCR aboutit souvent à des conclusions sur les similitudes des profils d'expression génique. Bien que l'ACP et le regroupement hiérarchique puissent donner des indications complémentaires sur les schémas de corégulation de l'expression génique, les deux techniques se concentrent sur les similitudes des profils d'expression génique. Cela limite les types d'hypothèses qui peuvent être trouvées dans les études exploratoires utilisant uniquement ces techniques. Pour élargir la portée des hypothèses générées dans les études exploratoires, une approche de l'analyse multivariée fondée sur des hypothèses a été récemment proposée24. Des algorithmes personnalisés et axés sur les hypothèses peuvent identifier des hypothèses biologiquement pertinentes que les techniques couramment utilisées pour l'analyse des données multivariées ne parviennent pas à mettre en évidence.
Matériaux
Pour continuer à lire, veuillez vous connecter à votre compte ou en créer un.
Vous n'avez pas de compte ?