Locked Nucleic Acid(LNA)およびMinor Groove Binder(MGB) / Eclipse Dark Quencher(EDQ)
Locked Nucleic Acid™(LNA®)およびMinor Groove Binder / Elipse Dark Quencher(MGB:EDQ)は、オリゴヌクレオチドの性能を高める修飾です。安定性を高め、標的配列との塩基対形成の特異性と親和性を向上させます。LNA®は、qPCRにおいて(BHQ™と併用されると)プライマーやプローブを強化し、アンチセンスオリゴヌクレオチド(ASO)などの治療法の有効性を高めます。一方、MGB:EDQは、クエンチャーが含まれており、qPCRにおいてプローブを特異的に強化します。
Locked Nucleic Acid™
LNA®は、2'-O, 4'-Cメチレンで架橋した新しいタイプの人工核酸です(図1)。この、3'-endoコンフォメーションの固定によって、リボフラノース環の柔軟性が制限され、非常に強固な二重らせん構造となります。これによって分析性能が高まり、用途の幅が広がります。

図1.LNA®モノマーとDNAモノマーの構造
PCRプライマー、qPCRプローブやその他の種類のオリゴヌクレオチド内のLNAは、水と標準的緩衝液に可溶性であり、ワトソン・クリックの塩基対形成の法則に従います1。
LNA®の利点
LNA®がオリゴヌクレオチドに組み込まれた場合には、DNA塩基のみの場合と比較して、次のようないくつかの利点があります。
- 熱安定性とハイブリダイゼーション特異性が高まります
- 遺伝子定量と対立遺伝子識別の正確性が高まります
- 困難なターゲットシーケンスに対して、より容易で柔軟に設計を行うことができます
熱安定性およびハイブリダイゼーション特異性の向上
LNA®をオリゴヌクレオチドに組み込むと、二本鎖の熱安定性が高まり2 、オリゴヌクレオチドの標的配列へのハイブリダイゼーションの特異性が向上します3。qPCRの場合は、これによってターゲット以外の配列への結合によるバックグラウンド蛍光が減少し、シグナル/ノイズ比(S/N比)が高まります。さらに、PCRプライマーまたはqPCRプローブの標的へのハイブリダイゼーションの向上によって、DNAオリゴヌクレオチドと比較して、融解温度(Tm)が、中間塩状態でのLNA®モノマー置換あたり最大8℃上昇する可能性があります4 (表1)。ハイブリダイゼーションにおけるこの向上によって、分析条件の範囲が大幅に広がり、より効果的なマルチプレックス解析が可能になります5。
遺伝子定量および対立遺伝子識別の正確性の向上
SNPを介して対立遺伝子を識別するオリゴヌクレオチドの能力は、LNA®塩基の組込みによって大幅に向上します6-8 (図2)。単一塩基ミスマッチの存在は、DNAオリゴヌクレオチドとその標的よりも、LNA®オリゴヌクレオチドとその標的の間の二本鎖形成により大きな不安定化作用を及ぼします。LNA®塩基を組み込んだ比較的短いオリゴヌクレオチドは、より長いDNAオリゴヌクレオチドと同じ温度で使用することができます。

図2.SNPジェノタイピング解析において、LNA両末端標識プローブはDNA両末端標識プローブより高い識別能を示します9。ピンク)LNA®変異プローブ(LNA®塩基3個を含む16 mer)による変異DNA解析。緑)DNA変異プローブ(25 mer)による変異DNA解析。赤)DNA変異プローブ(25 mer)による野生型DNA。紫)LNA®変異プローブ(LNA®塩基3個を含む16 mer)による野生型DNA。
困難なターゲットシーケンスのためのより簡単でフレキシブルなデザイン
LNA®は、ハイブリダイゼーション特性が向上し、それに伴いTmが上昇していることから、LNA®オリゴヌクレオチドは比較的短く合成することが可能であり、それによって、DNAオリゴヌクレオチドで生じる特定の設計上の問題が克服されます。特に、LNA®オリゴヌクレオチドは、ATまたはGCリッチ領域などの、従来は困難であった標的配列に対応するように設計することができます。例えば、ATリッチな天然状態のDNAオリゴヌクレオチドは、多くの場合、アンプリコン設計ガイドラインを満たすためには長さが30塩基以上である(場合によっては40塩基以上)必要がありますが、それでも性能が不十分なことがあります。LNA®オリゴヌクレオチドでは、LNA®塩基の選択的配置によって、わずか13~20塩基の長さでも十分な性能を発揮する、特異性が高く、短いオリゴヌクレオチドの設計が容易になります。また、比較的安定なG:Tミスマッチなどの、困難なSNPを標的とするオリゴヌクレオチドの設計も容易になります。
さらなる利点
LNA® PCRプライマーおよびqPCRプローブは、すべてのリアルタイムPCRやエンドポイントPCRに適合しています。専用の装置は必要ありません。
LNA®の用途
LNA®は、下記を含む、利用可能なすべての qPCR検出ケミストリーに組み込むことができます。
- SYBR® Greenプライマー
- 両末端標識プローブ
- モレキュラービーコン
- LightCycler® プローブ
- Scorpions® プローブ
下記に有用です。
- SNP検出
- 対立遺伝子の識別
- 病原体検出
- マルチプレックス
- ウイルス量の定量化
- 遺伝子発現解析
- 遺伝子コピー決定
以下の配列にも使用できます。
- アンチセンスオリゴヌクレオチド
- デコイオリゴヌクレオチド
- キャプチャープローブ
- アプタマー
- リボザイム
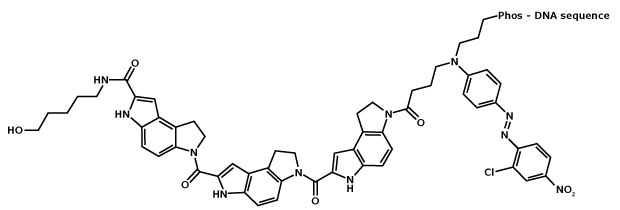
図 1.MGB:EDQの構造MGBであるCDPI3は、ジヒドロピロロインドールカルボキシレートのトリペプチドであり、そのブーメランのような湾曲により、B型DNAのminor groove(副溝側)の形状とぴったりと合うことにより、強力に結合することができます。
1. 溶液中のプローブは弱い発光を示す
2.加水分解による蛍光の放出
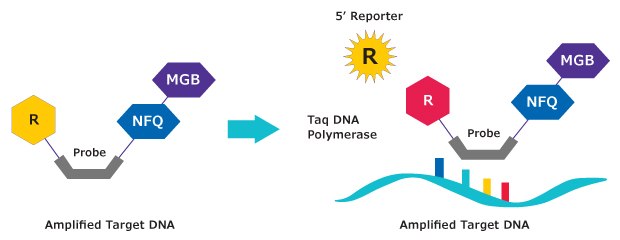
図 2.作用機序。MGB:EDQプローブは加水分解プローブです。ポリメラーゼによってプライマーが伸長し、特異的なDNA鋳型に結合します。加水分解によってプローブ/標的のハイブリッドからレポーターが放出され、蛍光が増強します。測定された蛍光シグナルは、標的DNAの量に正比例します。
MGBの利点
MGBは、いくつかの方法でqPCRの性能を高めます。
- 特異性の向上:MGBは、プローブと標的のハイブリダイゼーションの特異性を高めて、非特異的増幅の可能性を低下させます
- 感度の向上:MGBは、短いプローブの使用を可能にし、それによりqPCRアッセイの感度を高めます
- 融解温度調整:MGBにより、プローブのTm調整が可能になり、最適なTm値を持つqPCRプローブの設計に役立ちます
- バックグラウンドシグナルの低下:MGBは、バックグラウンド蛍光の低減に貢献し、シグナル・ノイズ比(S:N)が改善します
- 安定性の向上:MGBは、プローブ・標的ハイブリッドの安定性を高めて、プローブ分解の可能性を低下させます
- デザイン柔軟性:MGBの使用により、プローブデザインの選択肢が拡大し、効率的なqPCRアッセイの作成が容易になります
MGBで強化されたqPCRプローブは、信頼できる確実なqPCR結果を得るための貴重なツールです。
MGB:EDQの用途
MGB:EDQは、この qPCR検出ケミストリーに組み込むことができます:
- 両末端標識プローブ(6-FAM™、HEX™、TET™による標識、その他についてはお問い合わせください)
下記に有用です。
- ミスマッチの識別によるSNP検出
関連製品
参考文献
続きを確認するには、ログインするか、新規登録が必要です。
アカウントをお持ちではありませんか?