PCR/qPCR 데이터 분석
PCR/qPCR 정성 데이터 분석
기존의 PCR이 완료되면 아가로스 젤 또는 최근에는 모세관 전기영동 시스템을 통해 데이터를 분해하여 분석합니다. 일부 애플리케이션의 경우, SNP 유전자형 분석과 같이 분석에 사용된 엔드포인트 데이터로 qPCR을 실행합니다. 각각의 경우 엔드포인트 데이터는 PCR이 정체기에 도달한 후 정성적 분석을 제공합니다. 경우에 따라 엔드포인트 데이터를 분석하여 PCR 수율에 대한 반정량적 분석이 가능할 수도 있지만, 정량적 측정은 qPCR 및 정량화 주기 값(Cq)1 값 분석을 사용하여 더 자주 수행됩니다.
qPCR 데이터 분석
이 가이드 전체에서 PCR 또는 qPCR을 사용한 핵산 측정의 변동에 기여하는 요인을 강조했습니다. 이러한 각 요인을 최적화하여 반응에서 실제 유전자(표적)의 양에 가장 가까운 값을 제공하는 분석 결과를 도출해야 합니다. 이러한 프로세스의 결과로 각 샘플의 각 표적에 대한 Cq 값 세트가 생성됩니다. 이 장에서는 생물학적 스토리를 나타내는 신뢰할 수 있는 데이터를 제공하기 위해 이러한 Cq 값을 도출하고 분석하는 과정을 설명합니다.
정확한 Cq 값을 도출하기/h2>
기준선 보정
각 샘플의 각 대상에 대해 Cq 값이 결정됩니다. 서로 다른 기기와 연결된 여러 분석 패키지에는 Cq를 결정하는 대체 접근 방식이 있습니다(또한 Ct, Cp, 이륙점 등의 대체 이름을 사용하기도 합니다). 이러한 모든 알고리즘의 세부 사항을 자세히 설명하는 것은 이 가이드의 범위를 벗어납니다. 그러나 증폭 곡선을 기반으로 하는 qPCR 측정은 배경 형광에 민감합니다. 배경 형광은 플라스틱 제품 선택, 소멸되지 않은 프로브 형광 잔류, 시료 웰로 누출되는 빛, 주어진 마이크로시터 플레이트 웰에 대한 광학 검출의 차이 등 다양한 요인으로 인해 발생할 수 있습니다. 잘 설계된 분석에서는 증폭된 신호와 비교할 때 배경이 낮습니다. 그러나 배경 신호의 변화는 서로 다른 샘플의 정량적 비교를 방해할 수 있습니다. 따라서 기준선의 차이를 유발하는 배경 형광 변화를 보정하는 것이 중요합니다(그림 10.1).
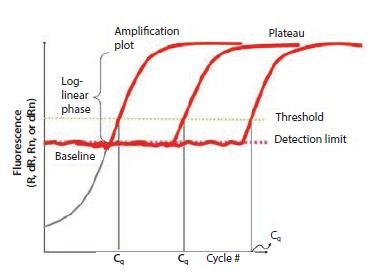
그림 10.1증폭 플롯의 구성 요소입니다. 이 그래프는 다양한 샘플의 사이클 수에 따른 형광의 증가를 보여줍니다. 임계값은 검출 한계보다 높게 설정되어 있지만 증폭 속도가 느려지는 정체기보다 훨씬 낮게 설정되어 있습니다.
일반적인 접근 방식은 초기 주기(예: 5~15주기 사이)의 형광 강도를 사용하여 배경 형광의 일정하고 선형적인 성분을 식별하는 것입니다. 그런 다음 이를 증폭 플롯의 배경 또는 기준선으로 정의합니다. 일시적인 효과로 인해 반응 안정화 인공물이 나타나는 경우가 많으므로 기준선 정의를 위해 처음 몇 사이클(예: 1~5사이클)은 피하는 것이 좋습니다. 기준선 보정에 사용되는 사이클이 많을수록 기준선 변화의 선형 성분의 잠재적 정확도가 향상됩니다. 많은 기기 소프트웨어 패키지에서 기준선 정의에 고려할 주기를 수동으로 설정할 수 있습니다. 사용자는 이러한 기능을 살펴보고 기본 설정에 대한 유혹을 강력히 거부해야 합니다.
기준선 설정의 효과에 대한 예는 그림 10.1에 나와 있습니다. 그림에서 볼 수 있듯이 정확한 기준선 설정에 따라 Cq 값과 증폭 플롯의 겉보기 모양이 영향을 받습니다. 이 예에서 C3으로 표시된 곡선의 기준선이 수동으로 잘못 조정되어 사이클 5에서 사이클 31까지의 데이터에서 계산된 기준선 주기가 잘못되었습니다. 이로 인해 곡선이 0 기준선 수준(그림 10.2A)에서 Cq가 28.80이 되는 딥 블로우 현상이 발생합니다. 이를 수정하기 위해 원시 데이터인 R을 보고 선형 배경의 마지막 주기(증폭 전 마지막 주기)를 식별합니다. <그림 10.2B>에서 이것은 사이클 22로 볼 수 있습니다. 기준선은 사이클 5와 사이클 22 사이에서 0으로 올바르게 설정되고(그림 10.2C) 증폭 플롯이 보정됩니다(그림 10.2D). 보정된 Cq는 26.12입니다. 따라서 기준선을 잘못 설정했을 때와 올바르게 설정했을 때의 Cq 값 사이에 상당한 차이가 있음을 알 수 있으며, 이는 올바른 기준선 설정이 데이터 분석의 중요한 요소임을 보여줍니다.
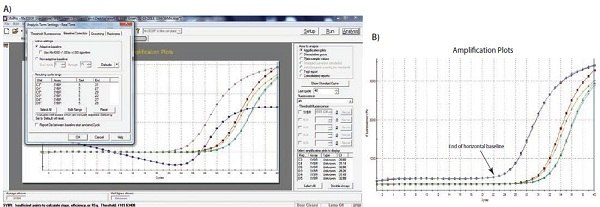
그림 10.2A-B. A)기준선 설정이 잘못되었을 때 데이터가 정규화된 형광 수치가 0 이하로 떨어지는 일반적인 예(파란색 증폭 플롯). B) 선형 기준선의 한계와 데이터에 결함이 없음을 보여주는 동일한 증폭 플롯의 원시 데이터.
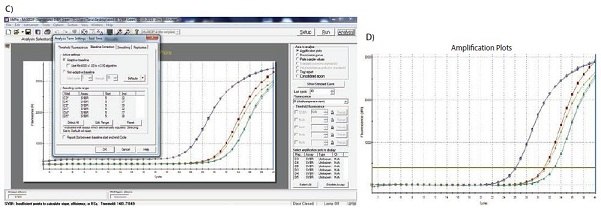
그림 10.2C-D. C)기준선의 시작과 끝의 한계는 적절한 소프트웨어 설정을 사용하여 정의합니다. D) 수정된 기준선 설정을 적용하면 양질의 데이터를 얻을 수 있습니다.
임계값 설정
일부 연구자들은 측정된 샘플의 증폭 효율과 목표 수량을 추정하기 위해 개별 증폭 플롯을 매핑하는 방법을 지지하지만2,3,4를 도출하는 가장 일반적인 방법은 임계값을 사용하는 것입니다. 이 접근법이 널리 채택된 것은 임계값 방법이 간단하고 효과적인 정량화 방법이기 때문일 수 있습니다.
임계값 방법의 원리는 다음과 같습니다: qPCR 증폭에서 관련 형광 신호를 시각화하려면 신호가 기기의 검출 한계(따라서 기준선, 그림 10.1) 이상이 되도록 증가해야 한다는 것입니다. 이를 위해 필요한 사이클 수는 샘플 내 표적의 초기 시작 사본 수에 비례합니다. 따라서 원래 카피 수가 낮으면 신호가 기준선 이상으로 증가하기 위해 더 많은 사이클이 필요하고 카피 수가 높으면 더 적은 사이클이 필요합니다. 기준선은 시스템의 탐지 한계로 설정되기 때문에 기준선에서의 측정은 매우 부정확할 수 있습니다. 따라서 시스템이 감지할 수 있는 최소 형광의 강도로 측정하는 대신 더 높은 형광을 선택하고 인위적인 임계값을 도입합니다.
임계값 강도를 선택하려면 몇 가지 기본 원칙을 준수해야 합니다. 임계값은 주어진 대상과 비교하려는 모든 샘플에 대해 고정된 강도로 설정하는 것이 중요합니다. 단일 플레이트에 너무 많은 샘플을 넣을 수 없는 경우, 플레이트 간 대조군 역할을 하는 복제된 대조군을 포함하거나 표준 곡선 직렬 희석과 같은 플레이트 간 교정 방식을 채택해야 합니다. 이론적으로 임계값은 증폭 곡선의 로그-선형 위상 어디에서나 설정할 수 있습니다. 그러나 실제로는 배경 형광 기준선 드리프트, 정체기 또는 분석 효율의 차이로 인해 증폭의 로그-선형 위상이 방해받을 수 있으며, 따라서 더 높은 사이클에서 증폭 플롯 기울기가 달라질 수 있습니다. 임계값은 다음과 같이 설정하는 것이 좋습니다.
- 배경 형광으로 인해 증폭 플롯이 임계값을 조기에 넘지 않도록 배경 형광 기준선보다 충분히 높게 설정합니다.
- 고원 단계의 영향을 받지 않는 증폭 플롯의 로그 단계에서(이는 로그 뷰에서 증폭 플롯을 보면 가장 쉽게 알 수 있습니다. 그림 10.3A).
- 모든 증폭 플롯의 로그 위상이 평행한 위치에서
임계값 설정 과정은 그림 10.3에 설명되어 있습니다. 그림 10.3A에서는 증폭 플롯을 Y축 로그 스케일로 표시하여 증폭의 로그 위상을 시각적으로 확장하고 이를 증폭 플롯의 선형 부분으로 표시합니다. 임계값은 이 로그 위상 내에 있고 모든 증폭 플롯이 평행한 가장 높은 형광 강도(Y축 참조)로 설정됩니다. 그러면 눈금이 선형 보기(그림 10.3B)로 반환되어 임계값 설정 요건을 충족하는 가장 높은 설정이 표시됩니다. 또는 이 로그 단계의 하단에 임계값을 설정할 수도 있습니다(그림 10.3C 및 10.3D). 증폭 플롯의 로그 위상이 평행한 경우 샘플 간의 ΔCq는 임계값 설정에 영향을 받지 않습니다.
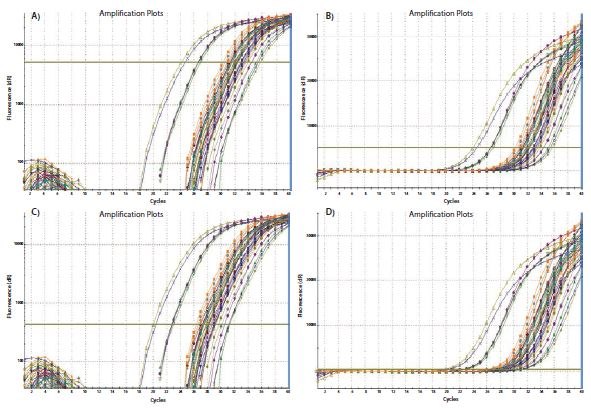
그림 10.3임계값 설정은 기록된 절대 Cq에 영향을 미치며 샘플 간의 ΔCq에 영향을 줄 수 있습니다. A). 데이터의 로그 대 선형 플롯을 사용하여 임계값은 가장 높은 형광 강도에서 설정되지만 증폭 플롯이 평행 로그 위상을 보이는 곳에서 설정됩니다. B). 임계값 설정은 A)에서 유지되며 선형 대 선형 플롯에 표시됩니다. C). 데이터의 로그 대 선형 플롯을 사용하여 임계값은 가장 낮은 형광 강도에서 설정되지만 증폭 플롯에 평행 로그 위상이 표시되는 위치에서 설정됩니다. D). 임계값 설정은 C)에서 유지되며 선형 대 선형 플롯에 표시됩니다. 각 경우에서 샘플 간의 ΔCq 값은 동일합니다.
증폭 플롯의 로그-선형 위상이 평행한 위치에서 임계값을 설정해야 한다는 요구 사항은 더 높은 주기의 데이터가 분석에 포함될 때 더욱 적절해집니다. 그림 10.3에서 데이터에 대해 설명한 임계값 설정 절차를 더 높은 Cq 데이터 세트에 대해 반복하고 그 결과를 그림 10.4에 표시했습니다. 결과 Cq 데이터는 표 10.1은 세 가지 임계값을 설정한 세 가지 증폭 플롯에 대한 ΔCq 값의 변동성을 설명하는 데 사용됩니다(그림 10.4). 증폭 플롯이 평행하지 않기 때문에 ΔCq 값과 각 샘플의 상대적 표적량 추정치는 임계값 설정에 따라 크게 달라집니다(그림 10.4).

그림 10.4.그림 10.3에서 수행 및 시연된 분석은 다른 데이터 세트를 사용하여 반복되었습니다. 이 경우 높은 Cq에서 반응 효율의 차이로 인해 증폭 플롯이 평행하지 않습니다. A) 및 B)의 가장 낮은 설정은 C) 및 D)의 가장 높은 설정과 다른 ΔCq 값을 초래합니다(표 10.1에 요약되어 있음).
qPCR 정량화 전략
정확한 정량화를 위해서는 정확한 기준선과 임계값 설정이 필수적입니다. 이들 각각을 설정하면 Cq 값이 생성되고 이를 정량화의 기준으로 사용합니다. 그런 다음 표준 곡선 또는 상대/비교 정량법을 사용하여 주어진 시료의 표적 수량을 결정합니다.
표준 곡선 정량
이름에서 알 수 있듯이 표준 곡선 정량법은 테스트 시료에서 표적의 수량을 결정하기 위해 표준 곡선을 사용해야 합니다. 따라서 시료에 대해 결정된 모든 수량은 표준 곡선에 할당된 수량에 상대적입니다. 이를 위해서는 모든 시료 반응 세트와 함께 추가적인 외부 표준을 실행해야 합니다. 표준 곡선에 대한 물질 선택은 시료와 표준의 분석 효율 차이로 인한 정량화의 잠재적 차이를 제거하는 데 중요합니다. 외부 표준의 프라이머 결합 부위는 표적과 동일해야 하며, 표적과 동일한 서열을 포함하고, 복잡성이 유사하며, 가능한 한 유사한 방식으로 처리되어야 합니다. 따라서 cDNA에서 표적의 농도를 측정할 때는 대조 시료의 직렬 희석에서 동일한 cDNA를 측정하는 것이 바람직합니다. 그러나 일부 연구의 경우 이를 방지하는 현실적인 이유가 있으므로 테스트 종과 관련이 없는 종의 gDNA를 인공 올리고뉴클레오티드 표준 또는 표준 서열을 지닌 선형화된 플라스미드에 추가하는 등 샘플 조건을 최대한 가깝게 재현하는 것이 중요합니다. 적합한 구조 또는 앰플리콘이 확인되면 직렬 희석의 표준 곡선이 생성됩니다. 표적에 대한 Cq가 각 표준에 대해 결정되고 농도 또는 상대 농도/희석 계수에 대해 로그 스케일로 플롯됩니다. 그 결과 표준 곡선이 생성되며, 이 곡선은 미지 시료의 증폭에서 도출된 Cq 값을 비교하여 테스트 시료의 농도를 결정하는 데 사용됩니다. 정량화를 위해 표준 곡선을 사용하는 경우, 표준 및 동일한 플레이트의 샘플에 대한 Cq를 결정하기 위해 임계값 설정을 일정하게 유지해야 합니다. 임계값은 플레이트마다 다를 수 있습니다.
상대/비교 정량
상대 또는 비교 정량에서는 다른 샘플에서 목표 서열의 농도 차이를 결정하는 요인으로 Cq 의 차이를 사용합니다. 표준 곡선 방법처럼 샘플당 표적의 양을 측정하는 것이 아니라 샘플 간의 배율 변화를 보여주는 데이터 세트로 이어집니다.
이 접근법의 원래 형태5에서는 모든 분석의 효율을 100%로 가정하여 Cq 차이가 1(ΔCq= 1)인 것은 표적의 2배 차이로 인한 결과였습니다. 표적 또는 관심 유전자(GOI)의 2배 변화를 확인하려면 데이터도 로딩 대조군(참조 유전자, ref; 데이터 정규화에 관한 논의는 다음 참조)을 참조해야 합니다.
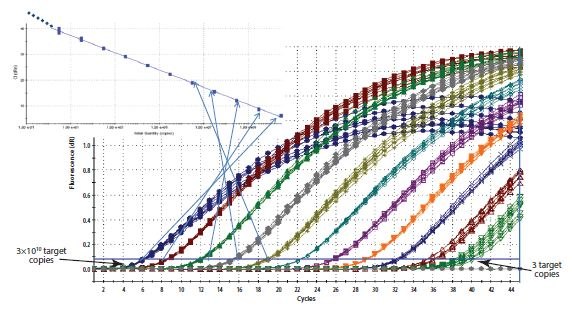
그림 10.5.표준 곡선 만들기. 희석 계열의 각 샘플에 대해 기록된 Cq는 상대 농도에 대한 로그 선형 눈금으로 그려집니다.
방정식 1에서, 참조 유전자에 대한 보정 후 두 샘플(B 대비 A)에서의 GOI의 비율은 다음과 같이 측정됩니다: 2(100% 효율적 반응 가정)를 GOI에 대한 Cq 값의 차이의 거듭제곱으로 나눈 값과 참조 유전자에 대한 Cq 값의 차이의 거듭제곱으로 나눈 값
.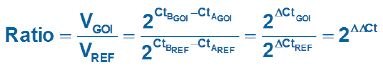
공식 1.오리지널(리박) 상대 정량화 모델.
그러나 분석 최적화 및 검증에 설명된 것처럼 반응의 효율은 상당히 다양하며 이는 데이터에 큰 영향을 미칠 수 있습니다. 따라서 반응 효율의 차이를 분석에 통합할 수 있도록 식 1의 가정을 해결했습니다(식 2)6. 이 경우 증폭 인자 2는 표준 곡선 분석에 의해 결정된 PCR의 실제 효율로 대체됩니다(분석 최적화 및 검증 참조).

방정식 2.효율성 적응형(Pfaffl) 상대 정량화 모델
효율 적응형(식 2) 상대 정량화 모델 사용의 예로, 표 10.2에 Cq 값 집합이 제시되어 있습니다. GOI의 효율은 1.8, 참조 유전자의 효율은 1.94입니다.
이 그림은 두 샘플에서 하나의 유전자를 단일 참조 유전자로 정규화한 후의 배율 차이를 측정해야 하는 매우 간단한 연구의 예입니다. 이 비율은 단일 참조 유전자로 보정한 후 샘플 1에 대한 샘플 2의 GOI의 배율 변화를 보여줍니다. 그러나 적합한 단일 참조 유전자를 선택하는 것이 종종 불가능하다는 것이 명백해졌으며, 따라서 정규화를 위한 보다 정교한 접근 방식이 제안되었습니다.
정상화
대부분의 PCR 기반 실험의 주요 목표는 샘플에 표적이 존재하는지(알 수 없음, UNK)에 대한 기본적인 질문을 해결하는 것입니다. 가장 간단한 수준에서는 젤을 실행하고 원하는 GOI의 존재 유무를 확인하기 위해 단편을 검사함으로써 이 질문에 답할 수 있습니다. 조각이 존재하는 경우, 조각 크기를 확인하면 양성 결과를 확신할 수 있습니다. 그러나 없는 경우 위음성 결과가 나올 가능성이 있습니다. 따라서 검사 분석을 반복하고 로딩 및 양성 PCR 대조군으로 사용하기 위해 최소 한 번 이상의 추가 PCR을 수행하는 것이 중요합니다. 범용 억제 제어 분석법인 SPUD(샘플 정제 및 품질 평가 참조)를 사용하면 음성 결과에 대한 확신을 뒷받침하는 데 사용할 수 있습니다. 또 다른 접근 방식은 참조 유전자에 특이적인 분석을 실행하는 것입니다. 기존에는 참조 유전자, GAPDH, 18S 리보솜 RNA 또는 β 액틴을 검출하는 PCR 분석을 GOI와 함께 실행하고 결과 단편을 겔에서 시각화했습니다. GAPDH, 18S 리보솜 RNA 및 β 액틴은 구성적으로 발현되므로 반정량 분석에서 로드 컨트롤로 사용되었습니다. 그러나 이러한 유전자가 실험 설계에 관계없이 모든 세포에서 동일한 농도로 보편적으로 발현되지 않는다는 것이 곧 명백해졌습니다. 따라서 예를 들어 유전자의 복제본 수 변이를 조사할 때 상대적인 핵산 농도, 일반적으로 cDNA뿐만 아니라 gDNA도 측정해야 할 때 안정적인 기준이 필요하게 되었습니다.
표준화는 실제 생물학적 변이를 조사하기 위해 기술적 측정치를 안정적인 기준으로 보정하는 프로세스입니다. 기술적 차이를 정규화하는 방법에는 여러 가지가 있으므로 특정 실험에 적합한 접근법을 선택하고 검증해야 합니다7. 부적절한 정규화 기법을 채택하는 것은 정규화를 전혀 하지 않는 것보다 전체 분석 프로세스에 더 해로울 수 있음을 인식하는 것이 중요합니다8.
시료 품질이 분석 정규화에 미치는 영향
시료 무결성과 순도가 qPCR 및 RT-qPCR을 통한 목표량 측정에 미치는 영향에 대해 자세히 논의했습니다(시료 정제 및 품질 평가, 시료 품질 관리 및 역전사, 리버스 트랜스크립션)를 참조하세요. 샘플의 억제제와 RNA 분해가 주어진 표적9 측정에 차별적인 영향을 미친다는 것이 입증되었습니다. 억제제는 모든 표적의 측정에 영향을 미치지만 분석 설계에 따라 그 정도가 다릅니다. 총 RNA의 분해는 전체 실험 설계에 따라 크게 달라지는 mRNA 및 miRNA10 측정에 영향을 미칩니다. 따라서 RT 반응에 대한 템플릿 농도의 영향과 정규화 후 샘플 품질이 데이터에 미치는 영향을 고려하는 것이 중요합니다. 정규화는 품질이 낮은 분석 또는 샘플의 영향을 상쇄하지 않습니다(분석 최적화 및 검증 참조).
표준화 접근법
이상적으로 정규화 방법은 qPCR 분석을 수행하는 데 필요한 다단계 프로세스 중에 발생할 수 있는 변동성에 대응합니다(그림 10.6). 그러나 프로세스의 어느 한 단계에서 정규화를 적용하면 이전 또는 이후 단계에서 각각 도입되었거나 도입될 기술적 오류 및/또는 편향을 제어하지 못할 수 있습니다. 정규화 방법은 상호 배타적이지 않으므로 여러 제어 방법을 조합하여 채택하는 것이 좋습니다11.
.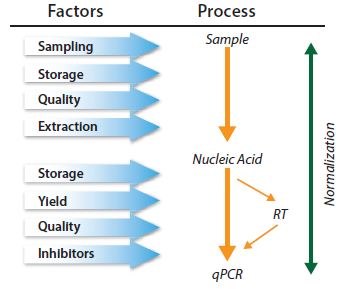
그림 10.6.qPCR은 다단계 프로세스이며 각 단계를 제어해야 합니다. 일련의 제어 내에서 정규화를 고려해야 합니다.
표준화의 목적은 측정값을 참조할 수 있는 안정적인 기준점을 제공하는 것이므로, 표준화 계수의 선택은 실험 전반에 걸쳐 안정적인 측정값이어야 합니다. 이는 안정적인 기준 유전자이거나 세포 수, 조직 질량, RNA/DNA 농도, 외부 스파이크12 또는 전체 발현 유전자의 대표 측정값과 같은 대안 중 하나일 수 있습니다.
참조 유전자 선택
참조 유전자는 실험의 결과로 양이 변하지 않는 표적입니다. 관심 있는 염기서열의 복사본 수가 변할 수 있는 DNA 복사본 수 변이를 정량화할 때, 변하지 않는 것으로 알려진 대체 게놈 영역을 표적으로 삼아 측정값을 간단히 정규화할 수 있습니다. 이 방법을 적용할 수 있는 예로는 인간 상피 세포 성장 인자 수용체 2(HER-2) 게놈 증폭13을 측정할 때를 들 수 있습니다. HER-2 게놈 불안정성은 유방암의 예후 지표로, HER-2 증폭 상태를 정확하게 측정하는 것이 환자 관리에 중요합니다. HER-2 상태는 대조군으로 작용하는 다른 유전체 표적과 HER-2의 사본을 비교하여 qPCR로 측정할 수 있습니다.
유전자 발현을 측정할 때 참조 유전자는 실험 결과 mRNA 농도가 변하지 않는 표적입니다. 예를 들어, 세포 단층에 유사 분화 화합물을 첨가한 후 유전자 X의 발현에 미치는 영향을 측정하는 연구를 들 수 있습니다. 유전자 X의 변화를 측정하기 위해서는 기준점이 필요하므로 해당 유사 분화 물질의 영향을 받지 않는 것으로 알려진 다른 유전자(또는 유전자)도 측정합니다. 따라서 연구자는 GOI를 연구하기 전에 실험 절차에 영향을 받지 않는 mRNA 표적을 찾아야 하는 즉각적인 과제를 안게 됩니다. 이러한 참조 유전자의 검증 과정은 GOI의 정확한 측정을 위해 필수적입니다. 정규화에 가장 널리 사용되는 접근 방식은 이 과정을 무시하고 유전자 발현 데이터를 검증되지 않은 단일 기준 유전자로 정규화하는 것입니다. 이 방법은 권장되지 않으며 MIQE 가이드라인1에 정면으로 위배됩니다. RT-qPCR에 의한 mRNA 정량화는 잘못된 기준 유전자 선택으로 인해 일상적으로 손상되었습니다. 프라이머가 이미 냉동실에 있거나, 과거에 노던 블롯에 사용했거나, 동료가 사용했거나, 다른 실험실에서 다른 실험에 사용했다는 이유로 참조 유전자를 사용하는 비교적 일반적인 관행을 따르는 것은 허용되지 않습니다. 특정 실험 시나리오에서 참조 유전자를 검증하여 해당 참조 유전자가 실험에 영향을 받지 않는지 확인해야 합니다. 이 검증을 수행하지 않고 참조 유전자가 실험의 영향을 받는다면 결과가 부정확할 수 있으며 후속 해석에서 무의미한 데이터8가 나올 가능성이 있습니다.
정상화를 위한 다양한 방법을 설명하는 다양한 과학 문헌7-14과 주어진 실험 시나리오에 가장 적합한 정규화 유전자를 식별하는 데 필요한 프로토콜을 설명하는 수많은 출판물이 있습니다. 과거에는 단일 또는 다중 참조 유전자를 선택할지 여부가 핵심 질문이었지만, 운영 비용이 낮아지면서 현재 모범 사례는 다중 참조 유전자를 측정하는 방향으로 바뀌었습니다.
안정적인 참조 유전자를 선택하려면 분석가가 테스트 및 대조 mRNA를 나타내는 샘플의 하위 집합에서 다수의 후보 mRNA 표적7 에 대한 qPCR의 안정성(보통 10~20개 유전자)을 평가해야 합니다. 전체 프로토콜은 부록 A, 프로토콜을 참조하고 REST15, GeNorm14, Bestkeeper16 또는 NormFinder17 같은 프로그램을 사용하여 다른 분석 방법과 함께 사용할 수 있습니다. 이 절차는 다음 섹션인 참조 유전자 안정성 분석에 자세히 설명되어 있습니다.
참조 유전자 안정성 분석
참조 유전자는 말 그대로 qPCR 상대 정량화 분석의 중심점입니다. 따라서 참조 유전자의 안정성은 전체 분석의 신뢰성을 위해 매우 중요합니다. 기준 유전자 발현이 샘플마다 달라지면 그 변화가 정량화 결과에 직접 전달되고 추가된 변동성이 원하는 관찰 가능한 생물학적 효과를 모호하게 만들거나 더 심하면 실제 관심 있는 유전자와 무관한 생물학적 효과가 완전히 인위적으로 나타날 수 있습니다. 이러한 이유로 참조 유전자의 변동성을 미미하게 만들고 생물학적 효과 측정을 가능한 한 유의미하게 만들기 위해 몇 가지 안전 조치를 따르는 것이 좋습니다.
가장 중요한 안전 조치는 하나뿐 아니라 두 개 이상의 참조 유전자를 사용하는 것입니다. 여러 참조 유전자의 발현을 평균화하여 정규화로 인한 기술적 변동성을 줄일 수 있습니다. 이는 작은 생물학적 효과의 측정에서 유의성을 높이는 데 유용할 수 있습니다. 그러나 더 중요한 것은 두 개 이상의 참조 유전자가 안정성을 유지하고 참조 유전자 중 하나의 발현 수준에 영향을 미칠 수 있는 예기치 않은 발생을 제어하기 위해 상호 제어 기능을 제공한다는 점입니다. 단일 참조 유전자를 사용하면 유전자 발현의 예기치 않은 영향을 분석에서 감지하지 못할 위험이 있습니다.
또 다른 안전 조치는 안정적인 참조 유전자를 식별하는 방법을 두 가지 이상 사용하는 것입니다. 다음은 참조 유전자 정규화의 여러 측면을 설명하기 위한 예로서, 동일한 데이터 세트에서 geNorm과 NormFinder 방법을 모두 사용할 경우의 이점을 포함합니다.
표 10.3는 이전에 EMBL과 진행한 워크샵에서 평가한 참조 유전자 후보 목록을 담고 있습니다. 샘플은 두 개의 다른 처리 그룹에서 인간 세포 배양에서 수집되었습니다. 이 데이터 세트는 참조 유전자 검증의 측면을 입증하는 데 사용됩니다.
NormFinder와 geNorm 알고리즘은 모두 다수의 참조 유전자 후보를 테스트하여 개별 참조 유전자 후보의 안정성을 평가하는 데 사용할 수 있다는 가정 하에 개발되었습니다. 예를 들어 모든 참조 유전자 후보가 안정적인 발현 수준에서 확률적으로 변화하는 경우 이 가정은 사실일 수 있습니다. 그러나 실제로는 그렇지 않을 수도 있습니다. 따라서 오해의 소지가 있는 결과를 피하려면 규제된, 특히 공동 규제된 참조 유전자 후보를 피하는 것이 현명합니다.
<표 10.3>에 표시된 참조 유전자 후보 목록은 서로 다른 기능 클래스에 속하는 유전자를 선별하여 유전자가 공동 조절될 가능성을 줄이기 위해 특별히 선택되었습니다. 주목할 만한 예외는 여기에는 두 가지 버전으로 존재하는 GAPDH입니다. 이 분석에는 영향을 미치지 않지만, 공동 조절이 의심되는 유전자를 여러 개 입력하지 않는 것이 가장 좋습니다.
첫 번째로 시연할 알고리즘은 geNorm입니다. 이 알고리즘은 분석된 참조 유전자 후보와 데이터 세트의 다른 모든 참조 유전자 후보 간의 쌍별 비교를 기반으로 M값이라는 유전자 안정성 측정값을 계산하여 유전자 안정성을 평가합니다. 이 절차는 반복적인 방식으로 수행되며, 이 예에서는 먼저 15개의 참조 유전자 후보 모두에 대해 절차를 수행하고 가장 안정성이 낮은 후보를 제거한 후 나머지 14개에 대해 이 과정을 반복하고 두 번째로 안정성이 낮은 후보를 제거하는 방식으로 두 개의 참조 유전자가 남을 때까지 반복합니다.
가장 안정적인 참조 유전자의 식별이 특히 어려운 경우가 있을 수 있습니다. 한 가지 경우는 모든 참조 유전자 후보의 성능이 좋지 않은 경우입니다. 또 다른 경우는 모든 참조 유전자 후보의 성능이 좋은 경우일 수 있습니다. 이 두 가지 경우를 구분하는 데 유용한 지침은 M값이 0.5 미만인 참조 유전자는 안정적으로 발현되는 것으로 간주할 수 있다는 것입니다.
두 번째로 시연할 알고리즘은 무료로 제공되는 참조 유전자 분석 패키지인 NormFinder입니다(부록 B, 추가 자료). 기본 알고리즘은 전체 및 하위 그룹에 대한 변이를 분석한다는 점에서 기준 유전자 안정성 평가에 대한 분산분석(ANOVA)과 유사한 접근 방식을 취합니다. 이 방법의 한 가지 장점은 얻은 측정값이 유전자 발현 수준과 직접적으로 관련이 있다는 것입니다. 따라서 Cq 단위의 표준편차 0.20은 특정 참조 유전자 후보의 복제수 발현 수준에서 약 15%의 변이를 나타냅니다.
편의상, 이 데모에서는 두 분석 패키지 모두 GenEx(MultiD) 데이터 분석 소프트웨어를 사용하여 액세스하지만 독립 패키지로도 제공됩니다(부록 B, 추가 리소스).
<그림 10.7>에 표시된 막대 다이어그램은 두 알고리즘을 사용하여 각각의 안정성 측정에 따라 순위가 매겨진 참조 유전자를 보여줍니다. 또한 NormFinder의 누적 표준 편차를 보여주는 그래프는 최대 3개의 최상의 참조 유전자를 조합하면 안정성이 향상될 수 있음을 나타냅니다.

그림 10.7.안정성 측정값을 보여주는 막대 다이어그램: geNorm의 M값과 NormFinder의 표준 편차. 또한 NormFinder의 누적 표준 편차를 보여주는 그래프는 최대 3개의 최상의 참조 유전자를 조합하면 안정성이 향상될 수 있음을 나타냅니다. 이 데이터 세트는 표 10.3에 표시된 참조 유전자 후보를 위해 설계된 분석에서 생성되었으며 두 가지 처리 그룹의 인간 세포 배양에서 측정되었습니다. 이 경우, 참조 유전자 안정성 알고리즘인 geNorm과 NormFinder가 최상의 참조 유전자에 대해 일치하지 않는다는 점에 유의하세요.
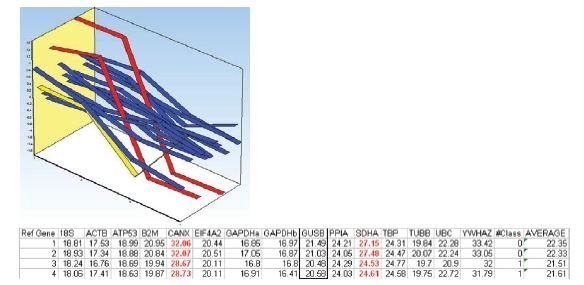
그림 10.8.각 처리 그룹에서 두 샘플의 기준 유전자 후보의 평균 중심 발현 프로파일입니다. 샘플 1과 2는 첫 번째 처리 그룹에 속하고 샘플 3과 4는 두 번째 처리 그룹에 속합니다. SDHA와 CANX의 발현 프로파일은 빨간색으로 표시되어 있습니다. UBC의 발현 프로파일은 노란색으로 표시되어 있습니다. 표에는 데이터 세트에서 측정된 Cq 값이 나열되어 있습니다.
표현 프로파일의 편차로 인해 SDHA와 CANX는 서로 다른 치료 대안에 의해 조절되므로 참조 유전자로 적합하지 않을 수 있습니다. 데이터 세트에서 이들을 제거하고 분석을 반복한 결과 두 알고리즘 간에 일치하는 결과가 나왔으며, 참조 유전자로 가장 적합한 것은 EIF4A2와 ATP53입니다(그림 10.9). 누적 표준 편차의 NormFinder 계산에서도 참조 유전자를 더 추가해도 안정성이 향상되지 않는 것이 분명합니다.
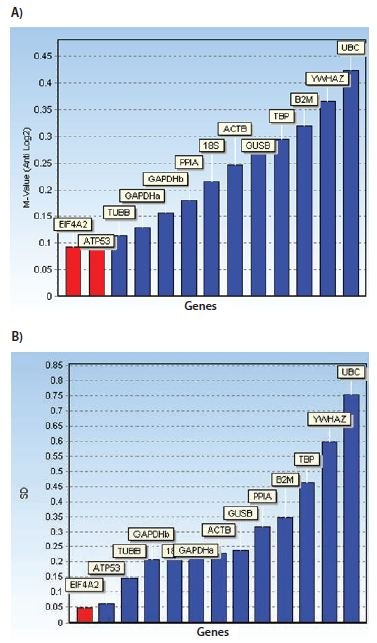
그림 10.9.발현 프로파일과 측정된 Cq 값(그림 10.8)을 검사한 결과, 적용된 분석에서 SDHA와 CANX가 공동 조절될 수 있다는 우려가 제기되었습니다. 이러한 공동 조절은 참조 유전자 안정성 알고리즘을 방해할 수 있습니다. 안정성 측정을 보여주는 막대 다이어그램: A) geNorm의 M-값 및 B) NormFinder의 표준편차. 데이터 세트는 그림 10.8에서 사용된 데이터 세트와 동일하지만 SDHA 및 CANX에 대한 데이터가 제거되었습니다. 이렇게 축소된 데이터 세트를 사용하면 참조 유전자 안정성 알고리즘인 geNorm과 NormFinder가 최상의 참조 유전자에 대해 일치한다는 것을 알 수 있습니다.
이 예시의 데이터 분석은 geNorm과 NormFinder를 병행하여 사용하면 공동 조절 참조 유전자 후보를 식별할 수 있으며, 이러한 유전자를 추가 연구에서 제거하면 단일 분석보다 더 확신을 가지고 채택할 수 있는 참조 유전자를 최종 식별할 수 있음을 설명합니다. 안정적인 참조 유전자를 식별하고 선택하면 데이터 분석의 보안이 강화됩니다.
대체 정규화 방법
참조 유전자에 대한 정규화는 분석 정규화를 위한 가장 일반적인 방법이지만, 이질적인 샘플 그룹에 있는 많은 수의 유전자를 비교해야 하거나 miRNA를 프로파일링할 때와 같이 이 접근 방식이 적합하지 않은 상황이 있습니다. 이러한 시나리오에서는 다른 전략을 채택해야 합니다.
조직 질량 또는 세포 수에 대한 정규화
정상화 인자로 사용할 세포 수 또는 조직 질량을 측정하는 것은 생각만큼 간단하지 않습니다. 세포 배양 실험은 세포 수를 기준으로 정규화하기가 비교적 쉽습니다. 그러나 처리를 추가하면 세포 형태에 영향을 미쳐 대조 배양과 비교할 때 세포 수와 발현된 총 RNA/유전자 비율을 복잡하게 만들 수 있습니다. 실험적 처리로 인해 여분의 세포 기질이 생성되어 핵산 추출 효율에 차이가 발생할 수 있습니다.
생물학적 조직은 피험자 내부 및 피험자 간에 매우 이질적일 수 있으며, 건강한 조직과 병든 조직을 비교할 때 더 큰 차이가 나타날 수 있습니다. 혈액과 같이 겉보기에 덜 복잡해 보이는 조직도 세포 수와 구성이 상당히 다를 수 있으므로 겉보기에 건강한 기증자18 간에 유전자 발현이 상당히 다를 수 있습니다.
핵산 정제에 사용되는 공정이 지연되면 측정된 RNA가 변경됩니다. 예를 들어 말초 혈액 단핵 세포를 처리하고 세포에서 RNA를 추출하는 과정이 지연되면 유전자 발현에 상당한 변화가 발생합니다19. 추출 절차의 기본이 되는 방법도 기술적 변동의 주요 원인입니다. 혈액 유래 세포를 샘플링하기 위해 선택한 분리 과정과 RNA 정제에 따라 겉으로 드러나는 유전자 발현 프로파일에 차이가 발생합니다20. 따라서 첫 번째 정규화 고려 사항은 모든 샘플에 대해 수집과 처리가 완전히 동일한지 확인하는 것입니다. 그런 다음 샘플 농도, 무결성 및 순도를 확신할 수 있도록 충분한 품질 관리를 수행하는 것이 중요합니다(시료 정제 및 품질 평가 및 관련 프로토콜은 Appendix A에 수록되어 있습니다.)
RNA 농도에 대한 정규화
최소한의 템플릿 농도(qPCR의 경우 DNA 또는 RT-qPCR의 경우 RNA)를 추정하는 것이 중요하며&에서 언급했듯이&.시료 정제 및 품질 평가에 따르면 핵산 농도 측정도 가변적이고 기술에 따라 달라지므로 모든 측정에 동일한 기기를 사용하는 것이 중요합니다.
총 RNA 농도를 측정할 때 대부분의 샘플은 rRNA로 구성되며 유전자 발현을 검사할 때는 관심 있는 mRNA, 유전자 발현 조절을 검사할 때는 sncRNA로 구성된 소량만 존재합니다. 즉, rRNA 농도가 소량 증가하지만 mRNA가 일정하게 유지되면 전체 RNA 농도가 증가합니다. 총 RNA 농도가 뚜렷하게 증가하려면 mRNA 농도가 상당량 증가해야 합니다. 따라서 rRNA 농도는 mRNA 농도의 신뢰할 수 없는 척도이지만, 많은 프로토콜에서 정확한 역전사를 보장하기 위해 동일한 RNA 농도가 필요합니다(역전사).
글로벌 유전자 발현에 대한 정규화
대량의 표적을 측정할 때 분석가는 전체 유전자 발현의 글로벌 평균을 추정하고 이 평균에서 벗어나는 조절된 RNA 서열을 식별할 수 있습니다. 이 접근 방식은 일반적으로 유전자 발현 배열의 정규화에 사용됩니다. 이는 참조 유전자를 사용하는 것에 대한 유용한 대안이며 많은 표적을 측정하는 경우 선호될 수 있습니다.
최근에 탐구된 또 다른 접근법은 많은 mRNA 내에 존재하는 내인성 발현 반복 요소(ERE)를 측정하는 것입니다. 많은 종에 이러한 반복 요소(영장류의 경우 ALU, 생쥐의 경우 B 요소)가 포함되어 있으며, 이를 통해 mRNA 분획을 추정할 수 있습니다. 이러한 표적 서열의 측정은 기존의 정규화 시스템9(Le Bert 등, 준비 중)과 같은 성능을 보이며 안정적인 기준 유전자 조합을 사용할 수 없는 복잡한 실험에 보편적인 솔루션 또는 대안을 제공할 수 있습니다.
miRNA 데이터의 정규화
아직까지 miRNA 범용 기준 유전자에 대한 보고는 없습니다. 따라서 정규화 시스템의 선택은 여전히 다소 경험적입니다. 가능한 경우, 마이크로어레이와 같은 게놈 전체 접근법을 통해 안정적인 불변 miRNA를 확인할 수 있습니다. 작은 핵 RNA(snoRNA)도 참조 유전자로 사용되었습니다. 글로벌 유전자 발현은 안정적인 참조 유전자를 알 수 없고 수백 개의 표적이 분석된 경우 miRNA 발현을 정규화하는 데 유용한 방법이기도 합니다21,22,23. 이 방법은 예를 들어 모든 miRNA를 다중화된 형태의 cDNA로 캡처하는 접근법을 사용하는 경우에 더 적합합니다, Exiqon 및 miQPCR 시스템(PCR 기술, 최신 혁신24에서 Castoldi 등 참조).
생물학적 및 기술적 복제본
정상화의 목적은 체계적인 오류를 피하고 최종 통계 분석을 위한 데이터 변동성을 줄이는 것입니다. 통계 분석을 위한 데이터 설정의 또 다른 중요한 측면은 데이터 복제본의 사용입니다.
생물학적 복제본은 통계 분석에 절대적으로 필요합니다. 통계적 유의 수준은 보통 5% 유의 수준 컷오프로 설정됩니다. 이러한 유의 수준에 가까운 생물학적 효과의 경우, 분석 유의 수준(1:20은 5%에 해당)을 결정하기 위해 최소 20개의 생물학적 복제본이 필요할 수 있습니다. 실제로 유의도25를 정확하게 추정하려면 최소 50배, 즉 생물학적 샘플 1000개 정도의 관찰 횟수를 기록해야 한다고 제안되었습니다. 물론 현실적인 제약으로 인해 이러한 수준의 생물학적 복제본은 거의 허용되지 않습니다. 또한 주어진 유의 수준을 충족하는 데 필요한 생물학적 복제본의 수를 정확하게 추정하는 것은 데이터의 변동성 수준에 따라 달라집니다. 그럼에도 불구하고 신뢰할 수 있는 결론에 도달하기 위해 필요한 생물학적 복제본의 수를 과소평가하는 것이 일반적인 실수라는 점을 인식하는 것이 중요합니다. 필요한 생물학적 복제본 수를 추정할 수 있는 좋은 근거를 마련하기 위해 분석의 고유한 변동성과 관찰 가능한 생물학적 효과의 잠재적 크기를 평가하기 위해 초기 파일럿 연구를 수행하는 것이 좋습니다26.
기술적 복제본은 통계 분석에 직접 사용되지 않습니다. 대신, 기술적 복제본은 샘플을 백업하고(기술적 처리 과정에서 일부 샘플이 손실된 경우) 데이터 정확도 평가를 개선하는 데 사용됩니다. 기술적 복제본은 기술적 처리 프로세스의 각 단계에서 정확한 측정값을 중심으로 확률적으로 변화한다는 가정이 사실이라면 데이터 정확도를 향상시킬 수 있습니다. 기술 복제본의 평균이 정확한 측정값에 더 가깝습니다. 기술 복제본 평균화의 효과는 미리 결정된 변동성, 즉 표준 편차를 1로 설정한 시뮬레이션 데이터 세트의 신뢰 구간 크기를 보면 설명할 수 있습니다. <표 10.4>에서 볼 수 있듯이 신뢰 구간은 기술적 복제본(샘플)의 수가 증가할수록 작아지며, 이는 정확한 측정값을 더 정확하게 추정할 수 있음을 나타냅니다. 또한 신뢰 구간이 좁아지는 현상은 기술적 복제본 수가 적을 때 가장 극적으로 나타납니다. 복제본 수를 2~3개로 늘리면 신뢰 구간이 8.99~2.48로 줄어들어 정확한 측정값 추정치의 정밀도가 3배 이상 향상됩니다. 복제본을 추가하면 측정 정확도 추정치가 계속 개선되지만 그 효과는 점점 감소합니다. 따라서 기술 처리의 변동성이 문제가 되는 경우에는 복제본보다는 삼중 복제본을 사용하는 것이 큰 이점이 될 수 있습니다.
RNA 추출, 역전사 및 qPCR 검출을 포함하여 샘플 처리 과정의 여러 단계에서 기술적 복제본을 수집할 수 있습니다. 여러 단계에서 기술적 복제본이 검출되면 중첩 실험 설계가 생성됩니다. 중첩 실험 설계를 활용하는 파일럿 연구는 기술적 처리 오류에 가장 크게 기여하는 샘플 처리 단계를 식별하는 데 도움이 될 수 있으며, 이 정보를 바탕으로 최적의 샘플링 계획을 계산할 수 있습니다27.
통계 분석 및 데이터 시각화
생물학적 데이터의 과학적 분석은 가설의 수립과 테스트에 중점을 둡니다. 가설을 세우려면 분석의 조건과 변수에 대한 자세한 이해가 필요합니다. 가설을 성공적으로 테스트하려면 기술적 변수를 최소화하면서 원하는 관찰 가능한 신호를 최대화하기 위해 신중한 실행과 적절한 실험 설계가 필요합니다. 이러한 맥락에서 탐색적 연구와 확증적 연구를 구분하는 것이 유용합니다(그림 10.10).
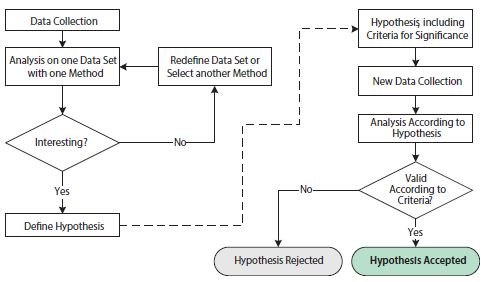
그림 10.10.탐색적 통계 분석과 확인적 통계 분석에 관련된 작업을 설명하는 순서도입니다. 그림의 왼쪽(점선 화살표 이전)은 탐색적 통계 연구의 작업을 보여줍니다. 그림의 오른쪽, 점선 화살표 뒤에는 확증 통계 연구의 작업을 보여줍니다.
탐색적 연구의 목적은 가설을 입증하기 위해 하나 또는 여러 가지 기법을 사용하여 데이터를 분석하는 것입니다. 하나 또는 여러 가설을 뒷받침하기 위해 데이터 집합을 재정의하거나 다른 분석 기법을 반복적으로 사용할 수 있습니다. 따라서 탐색적 연구는 과학적 질문의 세부 사항에 매우 유연하게 대응할 수 있습니다. 그러나 하나의 데이터 세트에 대한 가설 검증을 반복적으로 수행하면 통계적 결론을 약화시키는 문제가 발생할 수 있습니다. 이는 다중 검정으로 인한 것으로, 여러 개의 독립적인 가설이 있는 통계 테스트에서 양(+)의 유의성을 얻을 가능성이 더 높고, 기본 확률 분포가 동일하더라도 추가 가설을 테스트할수록 이러한 가능성이 증가한다는 사실을 말합니다. 따라서 오해의 소지가 있는 통계적 결과를 피하기 위해 탐색적 연구와 확증적 연구를 병행하는 경우가 많습니다.
확증적 연구의 요건은 훨씬 더 엄격한 통계적 기준을 기반으로 합니다. 첫째, 유의성 기준을 포함한 연구 가설은 데이터 수집 전과 분석 전에 정의되어야 합니다. 또한 분석용 데이터 세트는 이 목적을 위해서만 수집된 것이어야 합니다. 탐색 연구의 데이터 세트는 본질적으로 제안된 가설에 유리할 수 있으므로 확인 연구에서 데이터 세트를 재사용하는 것은 통계적으로 올바르지 않습니다. 확증 연구의 최종 결과는 미리 명시된 기준에 따라 가설이 거부되거나 받아들여집니다.
통계 테스트
통계 테스트에서는 관찰된 현상이 무작위 확률로 일어났을 가능성을 분석합니다. 이를 귀무 가설28이라고 합니다. 귀무 가설에 따라 관찰된 현상이 드물다면 귀무 가설이 타당할 가능성이 낮다는 결론이 나옵니다. 귀무 가설은 기각되고 대체 가설이 유의미한 것으로 받아들여집니다.
관찰된 현상이 무작위 우연에 의해 발생했을 것으로 추정되는 가능성을 p
-값이라고 합니다.-값은 0에서 1 사이의 범위 또는 이에 상응하는 백분율 단위로 측정됩니다. 확인 연구의 통계적 기준에는 계산된
-값이 관찰된 현상에 대한 유의성을 나타내는 알파 컷오프가 포함됩니다. 일반적으로 5%의 알파 컷오프가 사용되지만, 연구 대상에 따라 원하는 기준과 필요한 기준에 맞게 조정해야 합니다.
다양한 가정과 다양한 목적에 따라
-값을 계산하기 위한 많은 알고리즘이 개발되었습니다. 일반적인 알고리즘은 학생의 t-검정입니다. 학생 t 검정에서는 두 데이터 그룹 간의 평균값 차이를 기반으로
-값을 계산하는 데 사용됩니다. 학생 t 검정의 주요 가정은 두 데이터 그룹이 독립적이고 정규 분포를 따른다는 것입니다. 학생 t-검정의 장점은 비모수 통계 검정29에 비해 강력하다는 것입니다. 학생 t 검정과 동등한 비모수 통계 검정으로는 가장 잘 알려진 비모수 통계 검정 중 하나인 윌콕슨 순위 합계 검정(Mann-Whitney U 검정이라고도 하며, 두 쌍의 그룹을 비교하는 데 사용되는 윌콕슨 사인-랭크 검정과 혼동하지 말 것)을 들 수 있습니다. 윌콕슨 순위 합계 테스트와 같은 비모수 통계 테스트는 데이터 집합 분포에 대한 사전 가정에 의존하지 않는다는 점에서 학생 t-테스트와 같은 모수 통계 테스트에 비해 장점이 있습니다. 정규 분포에 대한 콜모고로프-스미르노프 검정을 사용하여 학생의 t 검정 또는 비모수 검정 중 하나를 적용할지 여부를 결정할 수 있습니다
알고리즘 선택과 더불어값 계산 알고리즘에 입력되는 데이터 집합을 조작하여 데이터 집합에서 원하는 속성을 쉽게 관찰할 수 있도록 할 수 있습니다. 원시 데이터 조작 단계와 p>-값 계산 알고리즘 선택의 조합은 가설 모델 구축의 일부입니다.
통계 분석의 탐색 단계에서는 가설 모델을 구축하는 데 높은 수준의 자유도가 있으며, 이는 과학적 탐구의 중요한 부분입니다. 그러나 가설은 과학적, 통계적 접근 방식으로는 결코 증명되지 않습니다. 올바른 과학적 접근 방식은 귀무 가설을 세우고, 독립적인(가급적 새로 수집한) 데이터 세트를 사용하고, 확증 연구 순서도(그림 10.10)에 따라 귀무 가설을 받아들이거나 거부하는 것입니다.
단변량 분석을 위한 시각화 기법
사용 가능한 분석 방법이 많은 것처럼 선택할 수 있는 데이터 시각화 기법도 많습니다. 단변량 데이터 분석의 경우 관련 오차 막대가 있는 간단한 막대 다이어그램이 적절한 시각화 기법입니다. 이것은 일반적이고 간단한 시각화 기법이지만, 강조할 만한 몇 가지 문제가 있습니다. 첫째, 오차 막대는 데이터의 고유한 변동성(표준 편차, SD) 또는 평균값이 결정된 정밀도 등 다양한 변동성의 원인을 나타낼 수 있습니다. 둘째, 평균값이 결정된 정밀도는 다양한 방식으로 설명할 수 있지만 궁극적으로 데이터의 내재적 변동성과 샘플 수(N)의 조합에 따라 달라지며, 원시 형태로 평균의 표준 오차(SEM, 식 1)라고 합니다:
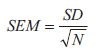
공식 1.SEM
그러나 SEM은 매우 직관적인 측정값이 아니며 서로 다른 실험의 SEM을 의미 있는 방식으로 비교하는 것은 간단하지 않습니다. 추정 평균의 정밀도를 설명하고 통계적 유의성을 그래픽으로 표시하는 더 널리 사용되는 방법은 신뢰 구간(CI, 식 2)입니다.
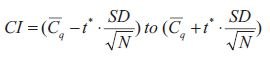
방정식 10-2.Cl
신뢰 구간 방정식에서 표준 편차(SD)와 표본 수(N)의 제곱근 사이의 비율로 SEM의 존재를 알 수 있으므로 신뢰 구간이 SEM에 기반하고 있음을 알 수 있습니다. 신뢰 구간의 하한은 평균에서 SEM에 t 분포의 백분위수를 곱한 값을 빼서 구성됩니다. 신뢰 구간의 상한은 평균에서 SEM에 t 분포의 백분위수를 곱한 값을 더하여 구성됩니다. 신뢰 구간의 신뢰 수준은 임계값 t*와 관련된 신뢰 수준(일반적으로 95% 신뢰 수준)으로 설정됩니다.
그림 10.11는 각 실험 그룹 내 95% 신뢰 구간을 나타내는 오차 막대가 있는 막대 그래프로, 여러 약물 용량으로 치료한 후 여러 장기의 샘플에서 유전자 발현 예시의 평균 추정치와 관련된 불확실성을 강조합니다. 또한 대조군 샘플과 각기 다른 약물 용량 반응에 따른 세 가지 샘플 간의 유전자 발현 차이에 대한 t-검정 통계적 유의성
값이 별표 표기로 표시됩니다. 별표 1개는 0.05 미만, 별표 2개는 0.01 미만, 별표 3개는 0.001 미만인 값에 해당하는 것이 일반적입니다.
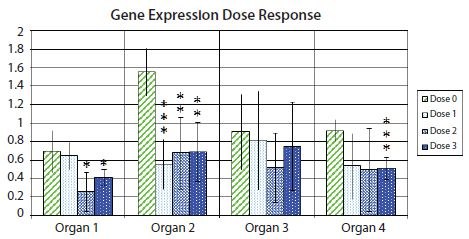
그림 10.11.각 장기 유형 내에서 가장 낮은 발현을 보인 샘플의 발현과 비교하여 한 쌍의 참조 유전자에 대한 관심 유전자의 2배 변화(log2) 발현. 막대 높이는 처리되지 않은(용량 0) 샘플 또는 세 가지 약물 용량(용량 1, 용량 2, 용량 3) 중 하나로 처리된 샘플 그룹에서 여러 샘플의 유전자 평균 발현을 나타냅니다. 오차 막대는 평균 표현식의 95% 신뢰 구간 추정치를 나타냅니다. 별표 하나는 처리된 샘플 세트의 평균이 처리되지 않은 샘플 세트의 평균과 비교하여 통계적으로 유의미한 차이가 5%, 별표 두 개는 통계적으로 유의미한 차이가 1%, 별표 세 개는 통계적으로 유의미한 차이가 0.1%임을 나타냅니다.
별표 표기가 p의 절대값을 숨긴다는 점을 고려할 때, p인 표 10.5의 예와 같이 절대값을 포함하는 것이 좋습니다. 그 이유 중 하나는 예를 들어 0.032의 p 값이 0.055의 p 값보다 약간 더 "유의미"하기 때문입니다. 이와 같은 경계선의 경우 데이터를 유의미한 것으로 분류할 때 사용할 컷오프를 정확하게 결정할 때 약간의 혼란이 발생할 수 있습니다. 현실적인 경우, p
값 0.051은 p 값이 0.049일 수도 있지만, 엄격한 (기본적으로 임의적이지만) 컷오프인 0.05를 적용하면 하나는 유의한 것으로 분류하고 다른 하나는 그렇지 않은 것으로 분류할 수 있습니다.그러나 평균 간 차이의 신뢰 구간을 활용하여 기존 막대 다이어그램의 단점 중 전부는 아니더라도 많은 부분을 피하는 막대 다이어그램 시각화의 변형이 있습니다24. 평균 간 차이의 신뢰 구간을 사용하면 생물학적 효과 크기와 데이터 변동성을 강조하는 동시에 관련 오차 막대와 함께 통계적 유의성을 직접 추정할 수 있습니다.그림 10.12는 그림 10.11에서 사용된 데이터의 평균 간 차이의 신뢰 구간이 있는 변형을 보여줍니다. 평균 간 차이가 0을 포함하지 않는 신뢰 구간은 p 값 컷오프에 해당하는 신뢰 수준에서 유의미한 결과에 해당합니다(그림 10.11> 및 표 10.5>에서 5%).
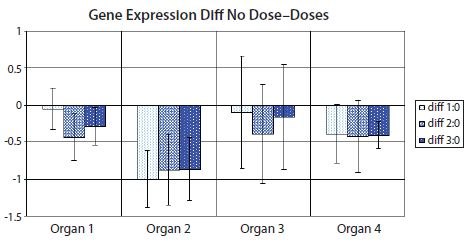
그림 10.12.그림 10.11의 데이터 세트에서 처리되지 않은 샘플 세트(용량 0)와 처리된 샘플 세트 중 하나(용량 1, 용량 2 또는 용량 3)의 평균 차이를 보여주는 막대형 다이어그램입니다. 오차 막대는 평균 차이의 신뢰 구간을 나타냅니다. X축을 가로지르지 않는 오차 막대는 해당 평균 비교가 t-검정에서 통계적으로 5%로 유의하다는 것을 나타냅니다. PCR 기술, 최신 혁신 -3rd ed. by Taylor and Francis Group LLC Books. 저작권 클리어런스 센터를 통해 서적/전자책에 재사용하는 형식으로 Taylor and Francis Group LLC Books의 허가를 받아 복제되었습니다.
다변량 데이터는 각 샘플링 단위에 대해 여러 변수에 대해 수집된 데이터입니다. 그림 10.11 및 그림 10.12에 사용된 데이터는 용량 및 장기 유형과 같은 변수에 따라 달라진다는 점에서 다변량 데이터에 해당합니다. 그러나 그림 10.11 및 그림 10.12의 통계 분석은 각 표현(막대)이 다른 변수의 고정 측정값과 비교하여 하나의 변수인 유전자 발현만을 나타낸다는 점에서 단변량 분석에 해당합니다. 다변량 데이터 분석 기법의 경우 계층적 클러스터링과 주성분 분석이 데이터 표현에 좋은 옵션입니다.
계층적 클러스터링
데이터를 특성화하는 가장 쉽고 유용한 방법 중 하나는 데이터를 분산형으로 그리는 것입니다(예: 측정된 Cq 한 유전자의 값을 다른 유전자의 해당 Cq 값과 비교하여 2D 플롯에서 생물학적 샘플 세트에 대해 플롯하는 것입니다.) 1차원 또는 2차원의 플롯은 사람의 눈으로 편리하게 시각화할 수 있습니다. 적절한 도구를 사용하면 3차원 플롯도 가능하지만, 고차원 플롯은 시각화하기가 훨씬 더 어렵습니다. 그러나 탐색적 연구의 경우 데이터 세트는 본질적으로 다차원이므로 전체 데이터 세트의 산점도는 비실용적일 수 있습니다. 예를 들어, qPCR 데이터 세트에는 여러 유전자 및/또는 여러 유형의 생물학적 샘플이 나타날 수 있습니다.
탐색적 연구에서 데이터를 특성화하고 시각화하는 인기 있는 대안적 방법은 산점도의 데이터 포인트 간 거리 측정값을 분석하는 것입니다. 유클리드, 맨하탄, 피어슨 상관관계 등 다양한 거리 측정값이 존재합니다. 계산 능력이 있다면 3차원보다 훨씬 더 높은 차원의 다차원 데이터에 대해서도 거리를 쉽게 계산할 수 있습니다. 응집 계층적 클러스터링의 경우 다음과 같은 반복 프로세스가 수행됩니다: 1) 가장 가까운 두 개의 개체를 찾아 클러스터로 병합하고, 2) 클러스터링 방법을 통해 새 클러스터를 새로운 개체로 정의하고, 3) 모든 개체가 클러스터30로 결합될 때까지 1)부터 반복합니다. 클러스터링 방법의 대안으로는 Ward의 방법, 단일 연결 및 평균 연결31이 있습니다. 덴드로그램은 종종 계층적 클러스터링 결과를 시각화하는 데 사용됩니다.
qPCR 데이터의 계층적 클러스터링 덴드로그램을 해석하면 종종 유전자 발현 프로파일 유사성에 대한 결론을 내릴 수 있습니다. 탐색 연구에서 이러한 유사성은 유전자 발현 핵심 조절에 대한 가설을 세우는 데 사용될 수 있으며, 이는 후속 확인 연구에서 받아들여지거나 거부될 수 있습니다. 계층적 클러스터링 덴드로그램의 장점은 유사성 관계를 명확하게 시각화할 수 있다는 점입니다. 반면에 유사성 측정에 중점을 두는 것은 가설 수립과 관련해서는 가설에서 유사한 표현 프로필이 중복 속성이 될 수 있으므로 제한적인 것으로 인식될 수 있습니다. 원하는 가설에 답하기 위해 특정 조합에서 서로를 보완하는 표현식 프로필 집합을 식별하는 것이 더 가치가 있을 수 있습니다.
주성분 분석
탐색적 연구에서 데이터를 특성화하고 시각화하는 또 다른 인기 있는 대체 방법은 전체 다차원 데이터 집합에 포함된 정보를 활용하고 원하는 속성을 선택하여 2D 또는 3D 플롯과 같은 저차원 분산형 차트에 투영하는 것입니다. 이는 주성분 분석(PCA)32,33,34,35을 사용하여 수행할 수 있습니다. 여기서 데이터 세트의 원래 좌표계(즉, qPCR로 측정한 발현 프로파일)는 새로운 변수(주성분: PC 또는 요인)가 구성되는 새로운 다차원 공간으로 변환됩니다. 각 PC는 원래 데이터 세트에 있는 피험자의 선형 조합입니다. 수학적 정의에 따르면, PC는 중요도에 따라 연속적으로 추출됩니다. 즉, 첫 번째 PC는 데이터에 존재하는 대부분의 정보(분산)를 설명하고, 두 번째 PC는 그보다 적은 정보를 설명하는 식으로 추출됩니다. 따라서 처음 두세 개의 PC 좌표(점수라고 함)를 사용하여 전체 데이터 집합을 2D 또는 3D 플롯으로 시각화하기에 적합한 편리한 작은 차원에 투영할 수 있습니다. 처음 두 개 또는 세 개의 PC를 사용하여 표현하면 데이터 세트에서 가장 많은 가변성을 설명하는 투영을 얻을 수 있습니다. 실험 설계 조건에 따른 분산은 체계적일 것으로 예상되는 반면 혼동 분산은 무작위적일 것으로 예상되므로 적절한 조건에서 이러한 표현이 필요할 수 있습니다.
앞서 계층적 클러스터링에 대해 언급한 것처럼, qPCR PCA의 해석은 종종 유전자 발현 프로파일 유사성에 대한 결론을 도출합니다. PCA와 계층적 클러스터링은 유전자 발현 공동 조절 패턴에 대한 상호 보완적인 인사이트를 제공할 수 있지만, 두 기법 모두 유전자 발현 프로파일 유사성에 초점을 맞추고 있습니다. 따라서 이러한 기법만으로는 탐색적 연구에서 발견할 수 있는 가설의 유형에 한계가 있습니다. 탐색적 연구에서 생성된 가설의 범위를 확장하기 위해 최근 다변량 분석에 대한 가설 중심 접근 방식이 제안되었습니다24. 가설 중심의 맞춤형 알고리즘은 다변량 데이터 분석에 일반적으로 사용되는 기법으로는 놓칠 수 있는 생물학적으로 관련된 가설을 식별할 수 있습니다.
자료
계속 읽으시려면 로그인하거나 계정을 생성하세요.
계정이 없으십니까?